III 型分泌系统 (Type III secretion system,T3SS) 主要是革兰氏阴性菌的分泌蛋白分泌到细胞外的运输途径,T3SS效应蛋白 (Type III secretion system Effector protein) 与革兰氏阴性致病菌致病机理有关。
通常用软件EffectiveT3 预测T3SS,通过其内部特定的计算模型对每条氨基酸序列进行评分,分值越高,可信度越高,选出评分高于阈值的序列,认为这些序列为III 型分泌系统效应蛋白。
EffectiveT3 的更新版本加强了 Effective 中 N 端信号肽的识别。对于更新,开发者收集了新的训练数据集,将来自 T3SEdb 的 504 个经过验证的分泌蛋白与其原始的训练数据结合在一起。新模型同样基于朴素贝叶斯分类器(Naive Bayesian Classifier),只是用了更多数据进行训练。在执行遗漏交叉验证测试(leave-one-taxon-out test )时,其精度为 0.87,与其之前的报告相当。
新模型现已嵌入到 Effective 中,也可供下载。在新模型中,朴素贝叶斯分类器对“secreted”类的默认最小分数为 0.9999。该默认值在网页上称为“selective”,而 0.95 称为“sensitive”。阈值也可以自由选择。
软件
主程序
依赖
- openjdk >=6
安装
1
2# 使用conda安装时依赖Pyton 2.7,3.5,3.6,需要首先创建相应版本的Python环境(自行创建)
conda install effectivet3数据库(modules)配置
记录一个深坑:程序默认在module的路径前加了一个”./module”路径,因此,虽然程序安装的过程中自动下载了modules,然而我们并没有办法调用它们,只能重新下载。用户每次运行软件前需要在当前目录下创建了module目录,并下载modules,然后将其存到module目录中。
1
2
3
4
5
6
7
8
9
10
11mkdir -p module
curl -o TTSS_STD-2.0.2.jar https://depot.galaxyproject.org/software/TTSS_STD/TTSS_STD_2.0.2_src_all.jar
curl -o TTSS_ANIMAL-1.0.1.jar https://depot.galaxyproject.org/software/TTSS_ANIMAL/TTSS_ANIMAL_1.0.1_src_all.jar
curl -o TTSS_PLANT-1.0.1.jar https://depot.galaxyproject.org/software/TTSS_PLANT/TTSS_PLANT_1.0.1_src_all.jar
curl -o TTSS_STD-1.0.1.jar https://depot.galaxyproject.org/software/TTSS_STD/TTSS_STD_1.0.1_src_all.jar
mv -f TTSS_STD-2.0.2.jar TTSS_ANIMAL-1.0.1.jar TTSS_PLANT-1.0.1.jar TTSS_STD-1.0.1.jar module
输入数据
包含蛋白序列的FASTA格式文件
运行软件
1 | effectivet3 -f F02_bin.1.faa -m TTSS_TTSS_STD-2.0.2.jar -t selective -o F02_bin.1.out -q |
- -f: 输入文件
- -m: 模型,可选”TTSS_ANIMAL-1.0.1.jar”,”TTSS_PLANT-1.0.1.jar”,”TTSS_STD-1.0.1.jar”和“TTSS_STD-2.0.2.jar”,建议用“TTSS_STD-2.0.2.jar”
- -t: 模式,”sensitive” and “selective”二选一,建议使用”selective”
- -o: 输出文件
- -q: 启动命令行模式
输出文件解读
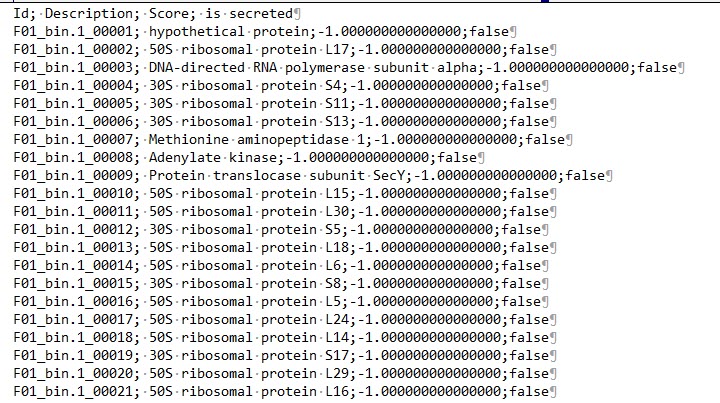
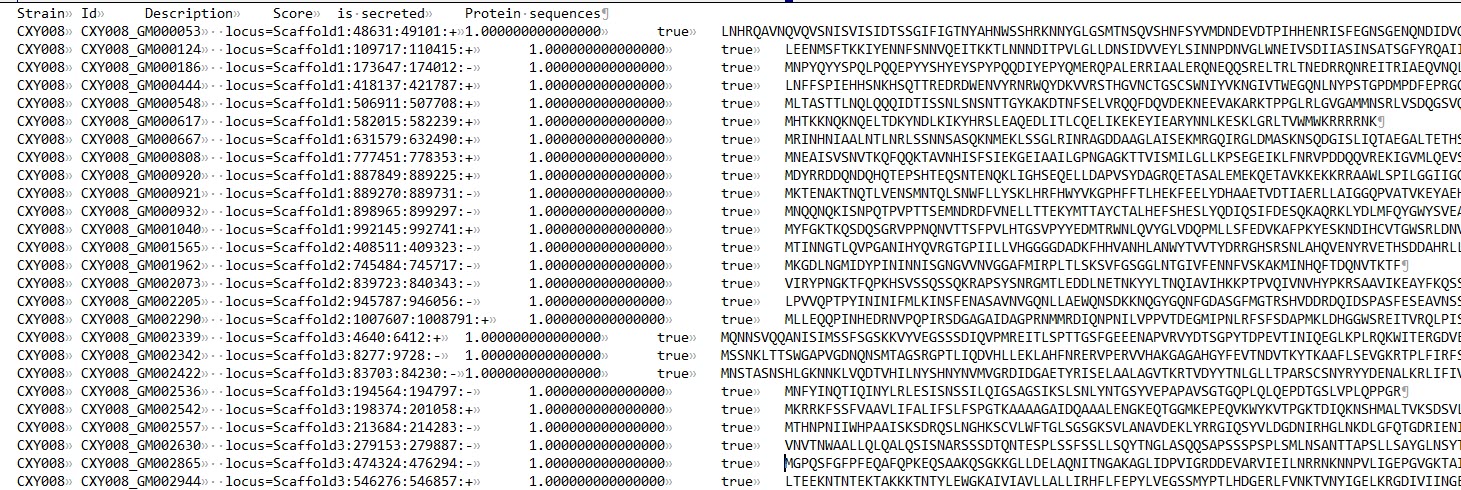
English: The table of results displays all query proteins sorted by prediction score. Effector classification (true/false) according to the applied threshold is shown in the last column.
简体中文: 结果包含了输入文件中所有的查询序列IDs,根据得分进行排序。一共包含4列,第一列为序列ID,第二列为序列描述,第三列为预测得分,第四列描述该序列是否为效应因子。各列间以英文的分号(;)分隔。
批处理与结果整合
脚本名:run_effectiveT3.pl
1 | #!/usr/bin/perl |
用法:将脚本与含有氨基酸序列的FASTA格式文件(后缀名为”.faa”,如果为其他,需要修改脚本第19、21和31行)放在同一目录下,在终端里运行如下命令(不需要事先配置数据库):
1 | perl run_effectiveT3.pl |
报错:Use of uninitialized value $seq in substitution (s///) at run_effectiveT3.pl line 47, <FAA> chunk .
原因:氨基酸序列ID/Header那一行的注释信息中含有”>“。该报错不影响结果的准确性,可以忽略。
结果解读
T3SS_result/strain_name.T3:(strain_name代表输入文件的名称)effectiveT3输出的原始结果,共4列,如前文所述。
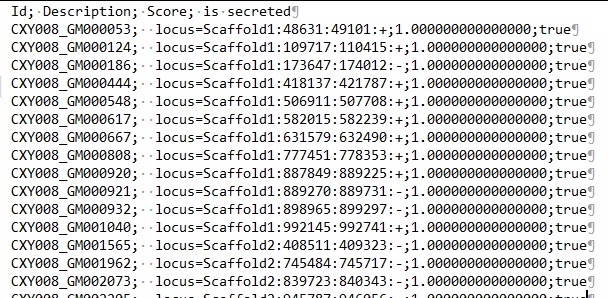
T3SS_result/T3SS.txt:包含了所有菌株的预测得到的效应因子,共6列,第一列为菌株名,中间的4列同上一个文件,最后一列为对应的氨基酸序列。
T3SS_result/T3SS.sum:记录所有菌株中序列总数、效应因子序列数和非效应因子序列数。

脚本获取
关注公众号“生信之巅”,聊天窗口回复“fb68”获取下载链接。
 |
 |
敬告:使用文中脚本请引用本文网址,请尊重本人的劳动成果,谢谢!