该文档可以帮助读者入门Linux系统,掌握常用命令和多种软件安装方式。此外还简述了全基因组测序技术,希望读者可以通过此文档初步了解生物信息学。
Linux初识
目录结构与路径
根目录
根目录(root)是系统最顶级目录,以“/”表示,其他目录均为根目录的下级目录。
家目录
家目录(home)是用户的最顶级目录,以“~/”表示,用户所有的目录均为家目录的下级目录。
绝对路径
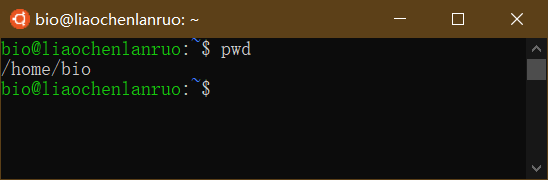
绝对路径指的是目录或文件的绝对位置,是从根目录开始的完整路径,如“/home/bio/”。可通过“pwd”命令获取当前目录的绝对路径。
相对路径
相对路径不需要从根目录开始,只要指定与当前目录的相对位置即可。
当前目录
当前所在的路径,以“./“表示。
上级目录
也称为父目录,以“../“表示向上一级的目录,以“../../”表示向上两级的目录,以此类推。
命名法则
- 文档与目录均以英文命名,可使用字母、数字和下划线;
- 文档与目录的名称不允许存在空格;
- 名称区分大小写。
终端工具
终端(terminal)是运行Linux命令的工具,类似于Windows的命令行工具。Linux各发行版均自带终端。

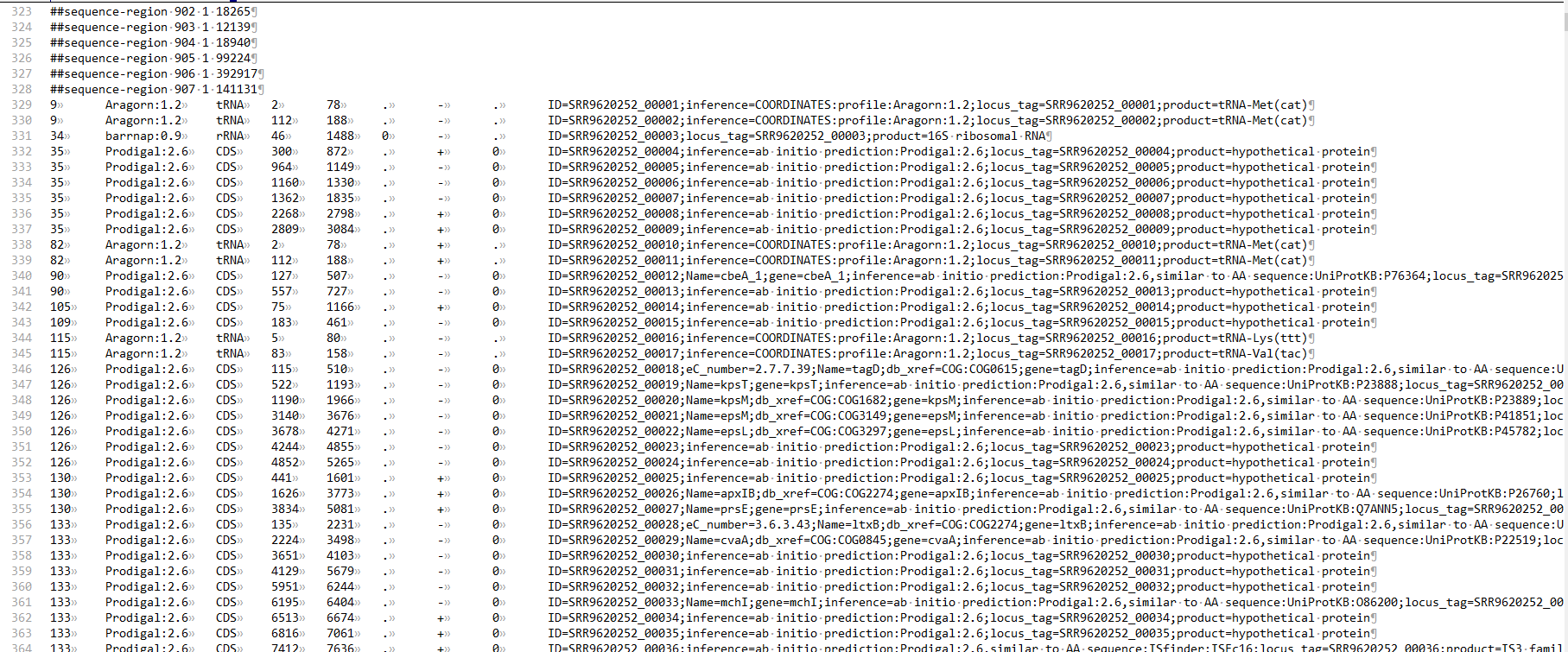
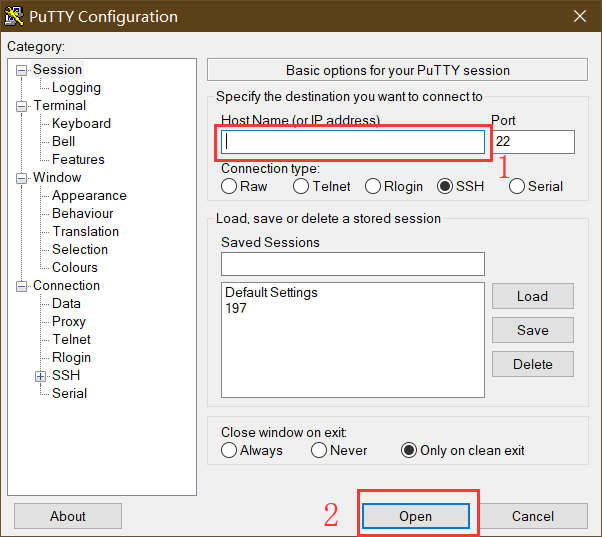
远程操控Linux服务器时,可以使用第三方的终端工具,如PuTTy软件。输入IP地址即可远程登录服务器运行命令。
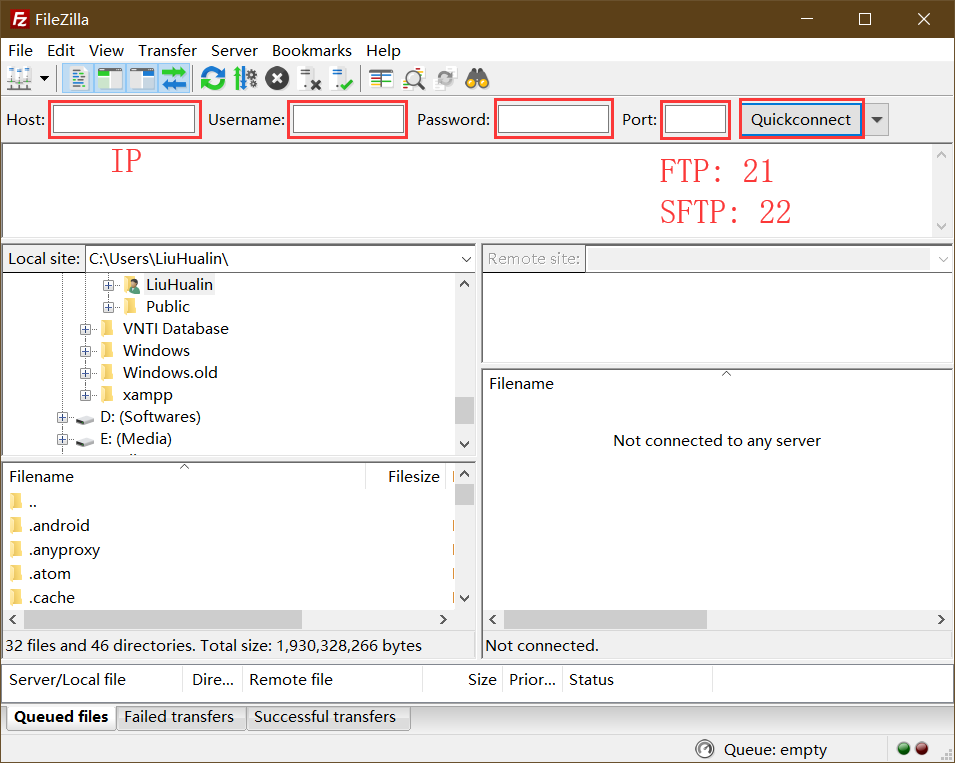
本地计算机与服务器之间的文件传输可以通过FTP软件实现,如FileZilla。输入服务器的IP地址、用户名、密码以及端口即可链接服务器。如果服务器采用的时FTP协议,则端口填写21,若采用的是SFTP协议,则端口设置为22。
常用的命令
pwd: 获取当前位置的绝对路径
1 | $ pwd |
mkdir: 创建目录
1 | $ mkdir |
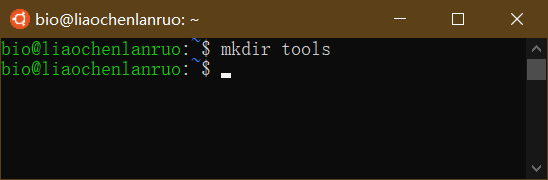
ls: 查看当前目录包含的内容
1 | $ ls |
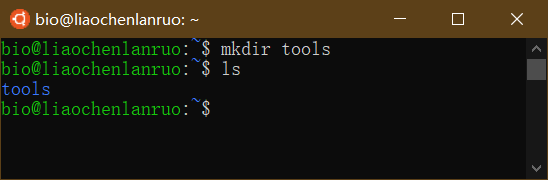
查看所有的目录和文件(包含隐藏的内容)
1 | $ ls -a |
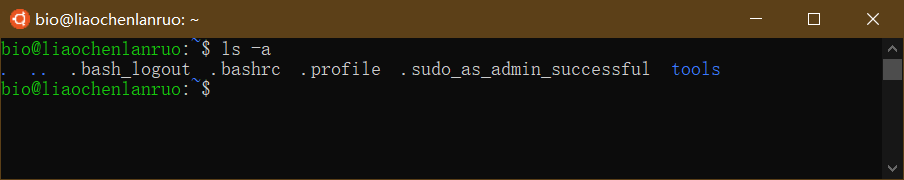
查看根目录所含内容
1 | $ ls / |
查看家目录所含内容
1 | $ ls ~/ |
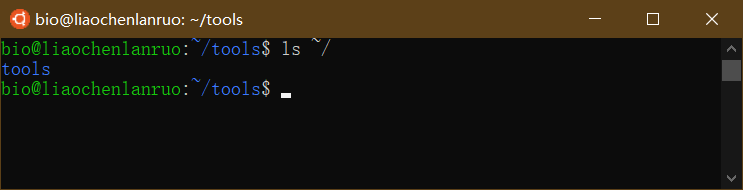
cd: 切换路径
进入“tools”目录
1 | $ cd tools |
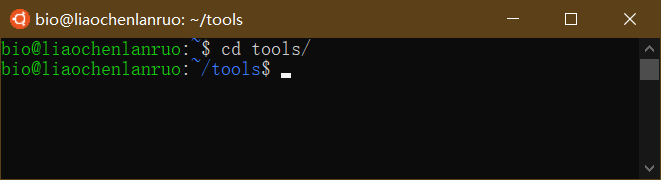
vim: 创建/编辑文档
以下所有操作均需在英文输入法状态下进行。首先创建一个新文档“example.txt”,并输入内容。
1 | $ vim example.txt |
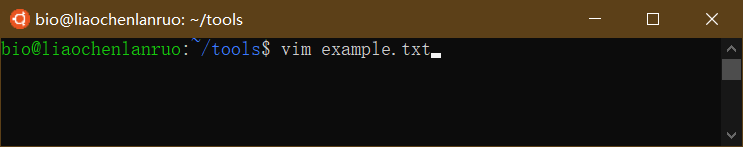

此时无法输入内容,需要按一下字母“i”键切换到输入模式。当左下角出现“–NSERT –” 字样时,可以输入文字。

输入相关的内容。
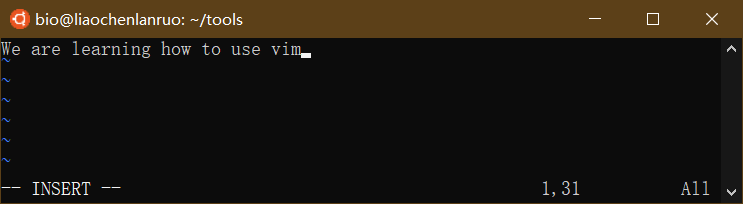
输入完毕时需要先按一下“ESC”键退出编辑模式,此时“–NSERT –” 字样消失。
按住组合键“shift + :”切换到vim操作模式,此时左下角出现一个“:”。
输入“wq!”保存修改并退出。
查看创建的文件是否在于目录下。
1 | $ ls |
cp: 复制目录或文件
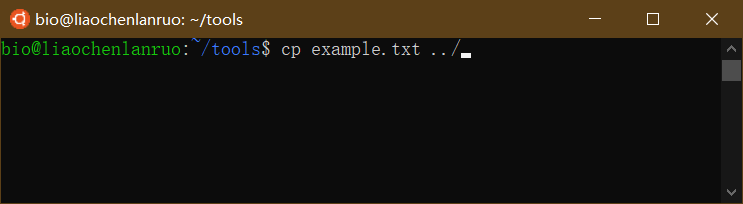
将创建的“example.txt”文档复制到上一级目录下。
1 | $ cp example.txt ../ |
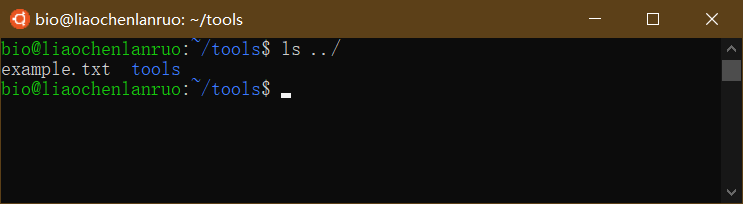
查看上一级目录下是否存在刚刚复制的文档。
1 | $ ls ../ |
rm: 删除目录或文件
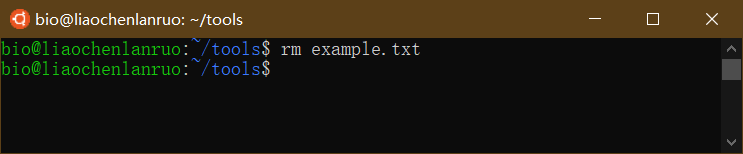
删除tools目录下的“example.txt”文档。
1 | $ rm example.txt |
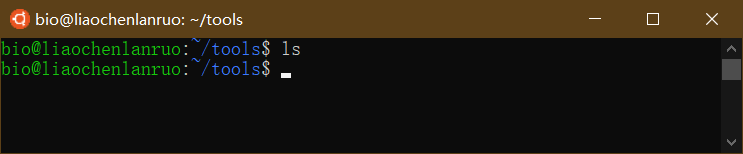
查看文档是否被删除。
1 | $ ls |
mv: 移动/重命名
将上一级目录下的“example.txt”文档移动到当前目录下。
1 | $ mv ../example.txt ./ |
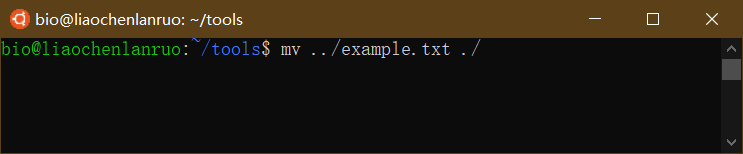
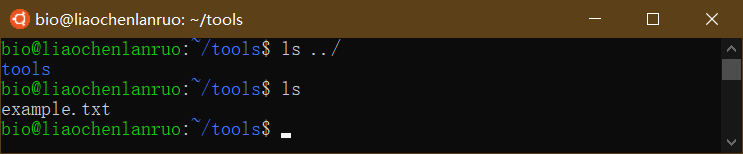
查看文档是否移动成功。
1 | $ ls ../ |
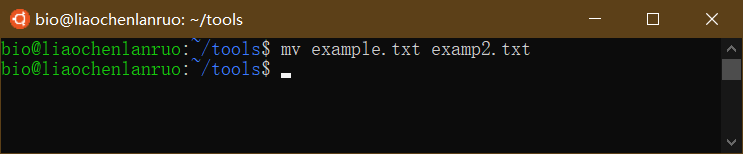
将“example.txt”文档重命名为“examp2.txt”。
1 | $ mv example.txt examp2.txt |
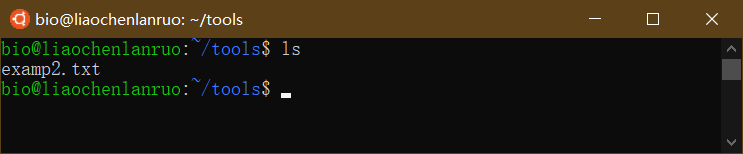
查看重命名结果。
wget: 下载
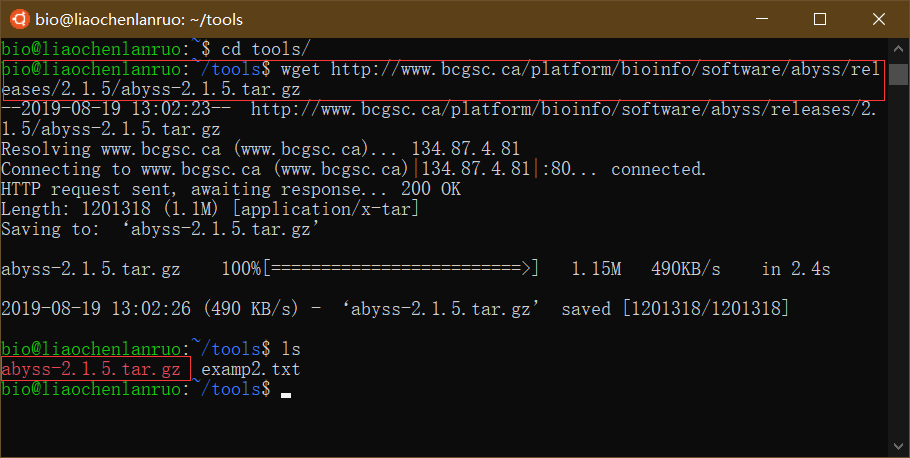
使用wget工具下载基因组拼接软件“AbySS”到tools目录下。
1 | $ wget http://www.bcgsc.ca/platform/bioinfo/software/abyss/releases/2.1.5/abyss-2.1.5.tar.gz |
tar: 压缩/解压缩
tar.gz格式的文件可用“tar zxvf”进行解压,将刚才下载的“abyss-2.1.5.tar.gz”解压缩。
1 | $ tar zxvf abyss-2.1.5.tar.gz |
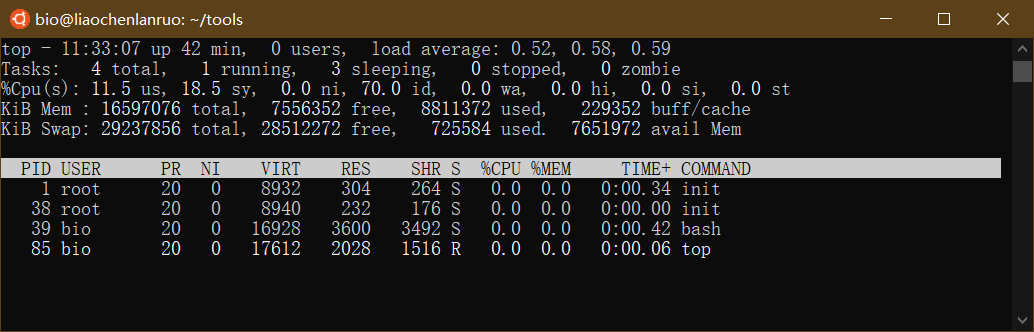
top: 查看系统进程
1 | $ top |
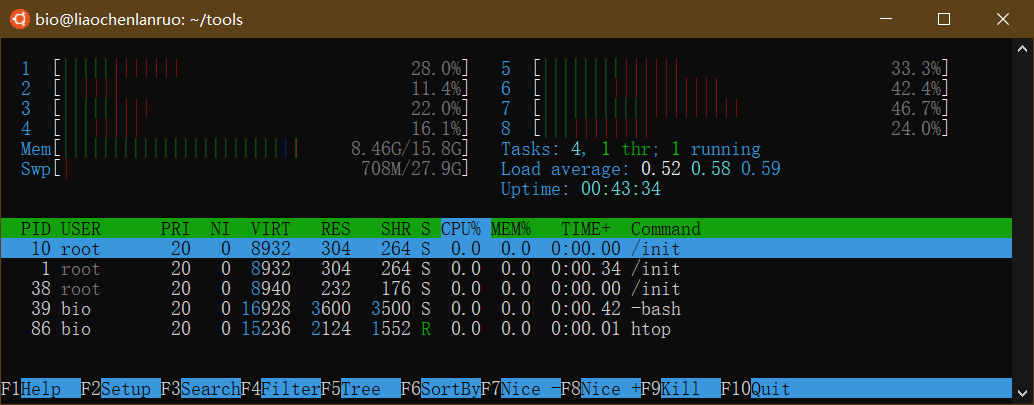
按字母键“q”退出。Ubuntu还带有另一个更加直观的查看系统进程的工具“htop”。
1 | $ htop |
环境变量
在软件安装的时候经常需要设置环境变量,所谓的环境变量就是告诉计算机软件的安装位置。存放环境变量的文件在用户的家目录下,为隐藏文件,可通过“ls -a”命令查看。
1 | $ ls -a ~/ |
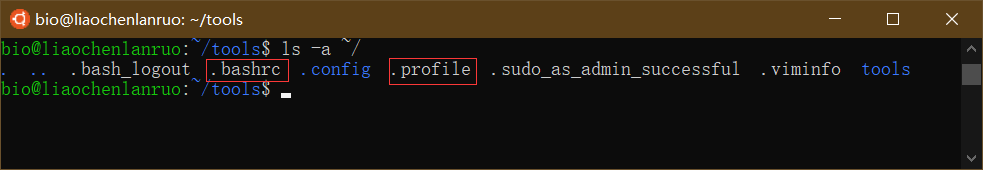
“.bashrc”和“.profile”均为环境变量配置文件,通常我们只需要编辑“.bashrc”。
软件安装
源码编译安装
源码安装适合于所有的Linux发行版以及macOS。以刚下载的“AbySS”基因组拼接软件为例演示源码编译安装,一共分三步:配置(./configure)、编译(make)和安装(sudo make install)。首先进入“AbySS”软件目录下,并查看目录中的文件,找到配置文件“configure”,根据“README.md”中的指示对软件进行配置。
1 | $ cd abyss-2.1.5/ |
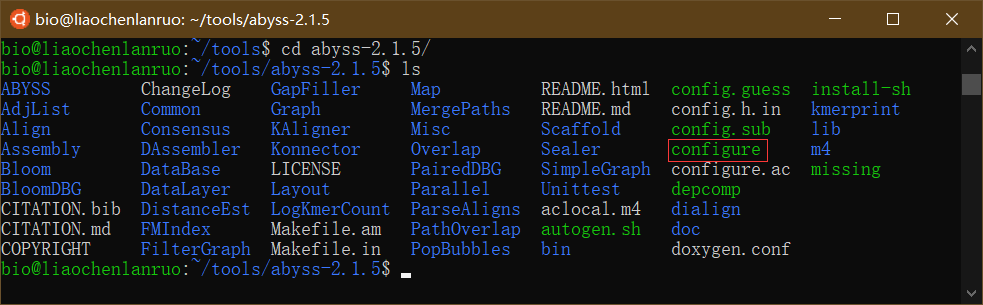
“./configure”表示运行configure进行安装前配置。
1 | $ ./configure |
进行编译
1 | $ make |
进行安装,需要“sudo”命令提供对系统目录的写入权限。
1 | $ sudo make install |
注意: 以上只演示了一般的安装方法,但是“AbySS”软件依赖其他的一些软件,需要先安装依赖包,最后安装“AbySS”,否则会安装失败。
通过包管理工具安装
不同的Linux发行版具有各自的软件包管理器。目前常用的Linux发行版主要是基于“RedHat”和“Debian”而制作的。
RedHat 系列的包管理器为“yum”,使用方法为在终端输入“sudo yum install -y 软件名称”。
Debian 系列的包管理器为“apt-get”,使用方法为在终端输入“sudo apt-get install 软件名称”。
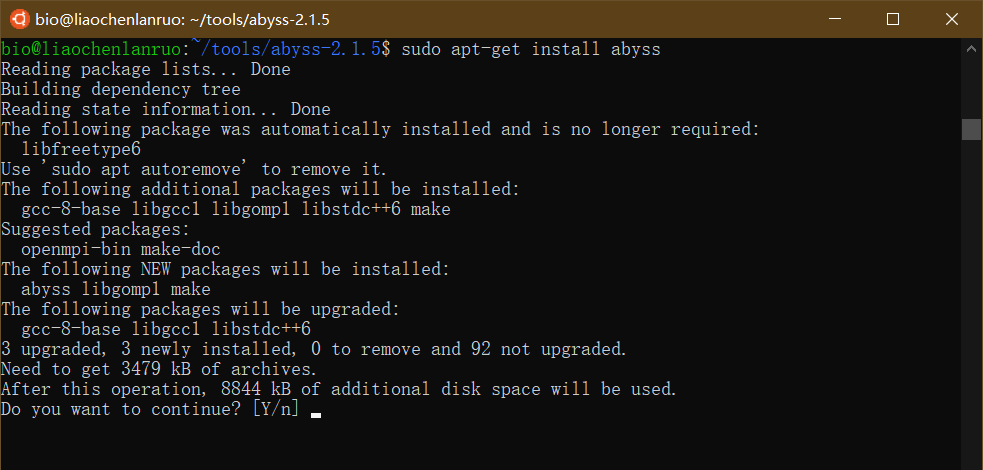
示例:通过apt-get在Ubuntu中安装AbySS软件,输入命令和密码后,根据提示输入“Y”并按回车键进行自动安装。
1 | $ sudo apt-get install abyss |
添加环境变量
以原核生物基因预测软件“Prodigal”为例演示。首先在Github上找到prodigal的源码,点击“Clone or download”,并按照图示点击链接右侧的图标以复制git地址。
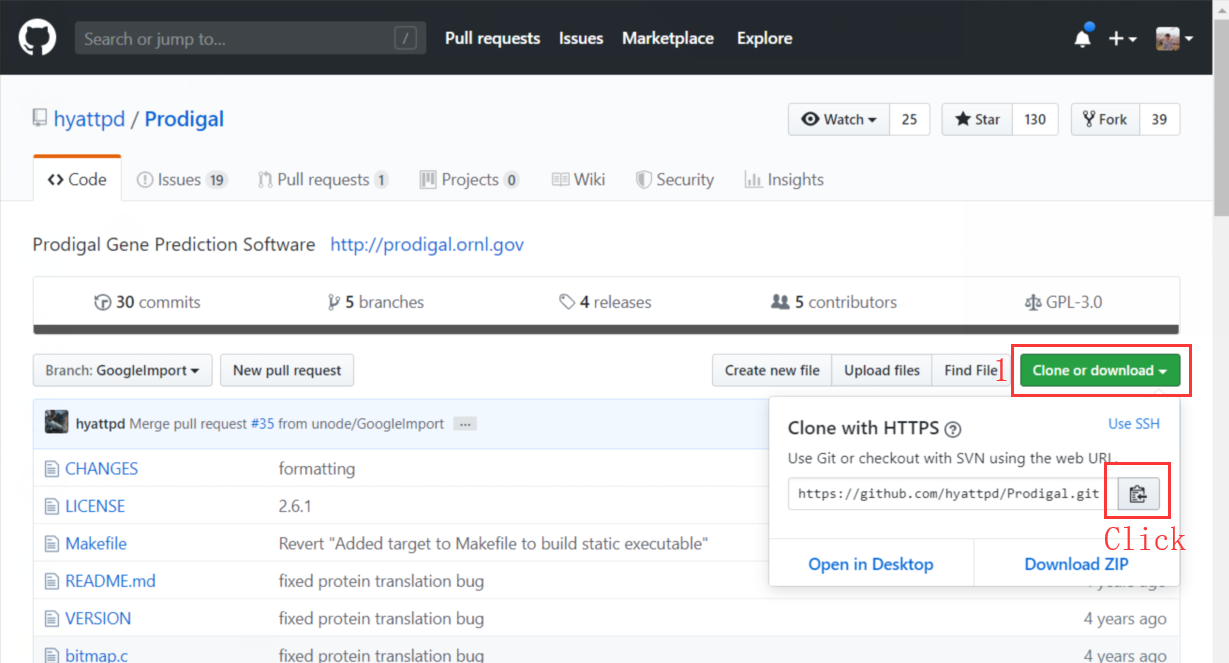
在终端中进入tools目录,并输入克隆命令将项目克隆到本地计算机。命令公式为“git clone link”。
1 | $ git clone https://github.com/hyattpd/Prodigal.git |
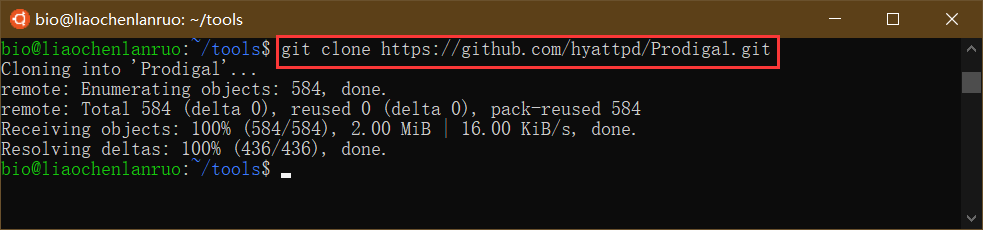
克隆完成后进入“Prodigal”目录。
1 | $ cd Prodigal |
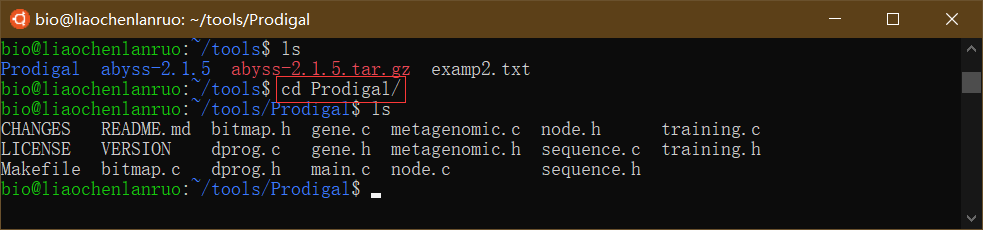
编译软件
1 | $ make |
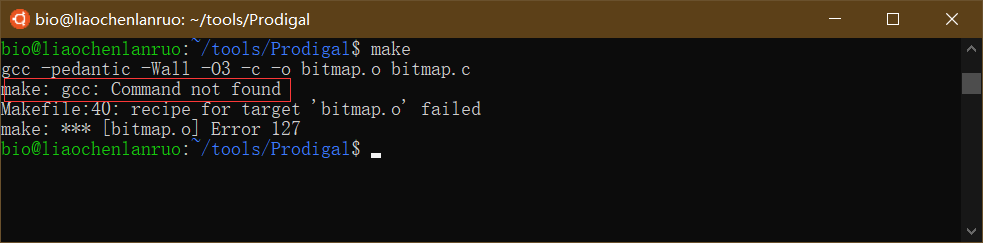
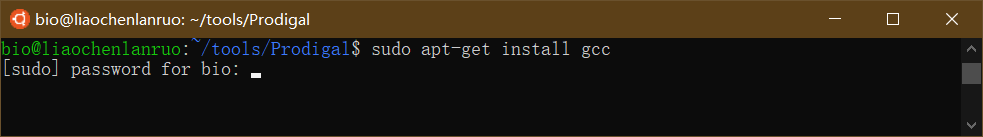
报错信息提示找不到gcc命令,因此需要首先安装gcc,输入命令后根据提示输入密码,直至安装完成。
1 | $ sudo apt-get install gcc |
重新编译prodigal
1 | $ make |
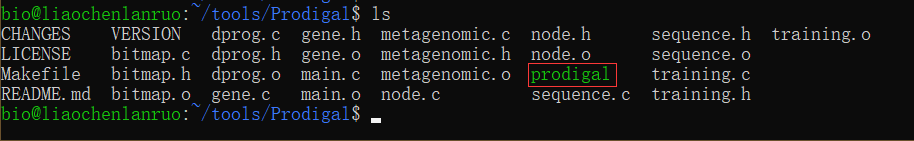
编译完成后得到了可执行程序,但是系统无法找到prodigal的路径,因此需要我们将其所在的路径加入到环境变量中。通过vim打开环境变量配置文件“.bashrc”,进入编辑模式。
1 | $ vim ~/.bashrc |
在文档末尾添加配置语句 “export PATH=$PATH:$HOME/tools/Prodigal” 。$HOME代表家目录,“$HOME/tools/Prodigal”代表prodigal可执行程序所在的目录。
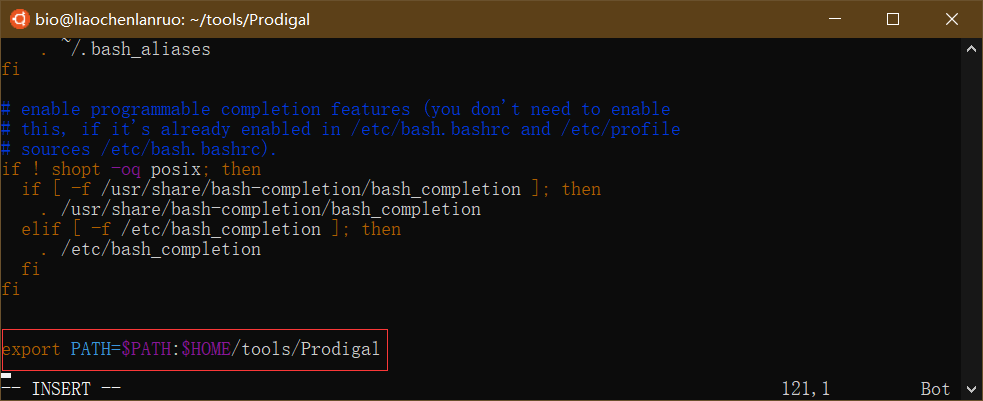
编辑完成后保存并退出。然后执行“source ~/.bashrc”命令刷新,通知系统“.bashrc”文档已经更改。
1 | $ source ~/.bashrc |

测试配置是否成功。
1 | $ prodigal -h |
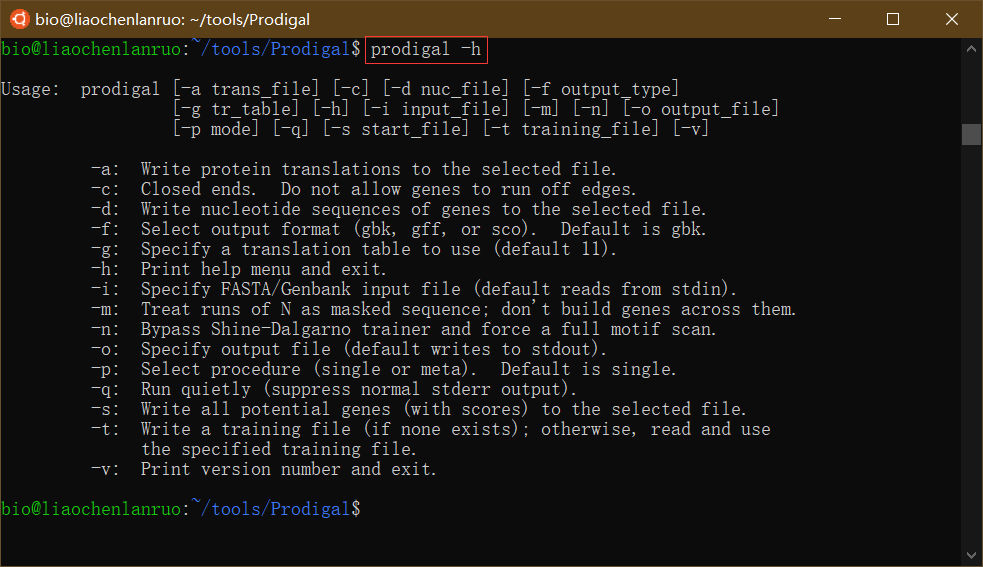
若要将其他软件加入到环境变量,只需在后面加入其他软件所在路径即可,各软件的路径间以英文“:“分割,不得有空格。下图为将多个软件加入到环境变量的示例。

创建软链接
软链接(Soft Link)相当于Windows系统中的快捷方式,可以将可执行程序的软链接存放至系统默认的环境变量之中,如“/usr/bin/”或“/usr/local/bin”之中。仍旧以刚编译好的prodigal软件为例,创建软链接的公式为 “sudo ln -s /home/bio/tools/Prodigal/prodigal /usr/local/bin/prodigal” ,根据提示输入密码完成创建。
1 | $ sudo ln -s /home/bio/tools/Prodigal/prodigal /usr/local/bin/prodigal |
通过“whereis”命令查看软链接是否创建成功。
1 | $ whereis prodigal |
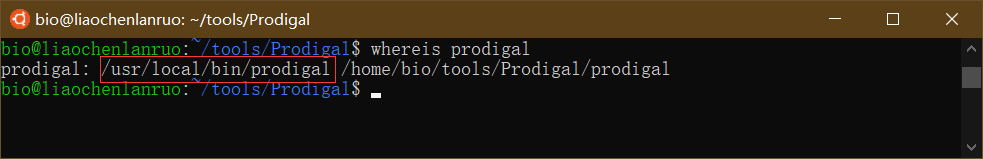
注意: 创建软链接时要输入绝对路径,否则会报错“Too many levels of symbolic links”。
通过Anaconda包管理器进行安装
Anaconda是一款比较易用的跨平台软件包管理器,Bioconda是conda的一个通道,专门管理生物信息学软件。通过conda安装软件时可以一键安装所有的依赖包,大大节约了时间并降低了安装难度。Bioconda目前有超过600个贡献者和500个成员,大部分生物信息学软件都被包含其中。用户可以到其官网搜索需要的软件是否被囊括其中。
(1)安装conda
此处,我们安装Miniconda,进入官网,选择适应自身系统及python版本的安装文件。
![]()
查看系统python版本
1 | $ python -v |
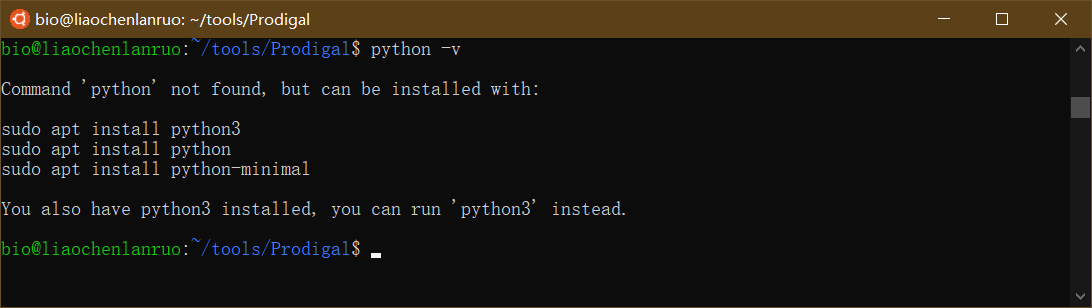
可以看出该系统已经安装了python3,因此下载Linux Python 3.7 64-bit (bash installer)。右键单击相应安装包获取链接,使用wget下载至tools目录下。建议用户安装Python 3,因为Python 软件基金会将于2020年元旦停止对Python 2的维护(https://pythonclock.org/)。
1 | $ wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh |
![]()
开始安装Miniconda
1 | $ bash Miniconda3-latest-Linux-x86_64.sh |
根据提示按“Enter”键查看license,并输入“yes” 按“Enter”继续,按“Enter”确认安装位置,miniconda被安装到家目录下的miniconda3目录中。最后输入“yes”,按“Enter”进行初始化。最后,通过“source ~/.bashrc”命令刷新。
![]()
(2)设置bioconda channel
在终端中输入以下三条命令添加channels:
1 | $ conda config --add channels defaults |
至此,bioconda配置完毕,可以通过conda安装生物信息学软件。下面通过conda安装mapping软件“bwa”。
1 | $ conda install bwa |
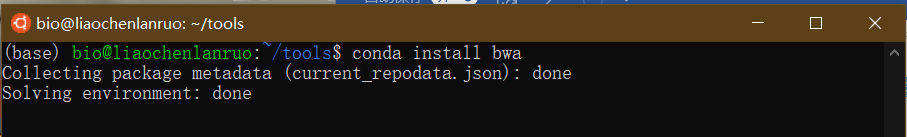
根据提示输入“y”完成安装。
MacOS相关操作
MacOS与Linux系统相似,基本命令相同,但是软件安装存在一些差异。
MacOS安装生物信息学软件
源码安装
源码安装方式与Linux安装方式一致。
创建软链接
配置方法与Linux一致。
环境变量
MacOS环境变量配置方法与Linux配置方法一致,但配置文件为家目录下的“.bash_profile”,即运行如下命令进行编辑。
1 | $ vim ~/.bash_profile |
编辑完成并保存后需要运行source命令。
1 | $ source ~/.bash_profile |
包管理器
MacOS的软件包管理器为Homebrew,可以在终端中通过以下命令安装Homebrew。
1 | $ ruby -e "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/master/install)" |
通过Homebrew安装mapping软件“bwa”。
1 | $ brew install bwa |
MacOS配置Anaconda
安装Miniconda
1 | $ wget https://repo.anaconda.com/miniconda/Miniconda3-latest-MacOSX-x86_64.sh |
添加Bioconda通道
1 | $ conda config --add channels defaults |
安装软件bwa
1 | $ conda install bwa |
现代测序技术
二代测序(“Next-generation” sequencing technology)
第二代测序技术的核心思想是边合成边测序(Sequencing by Synthesis),即通过捕捉新合成的末端的标记来确定DNA的序列。应用最广的技术平台主要为Illumina公司的产品。其优点为高通量、错误率低、成本低等。
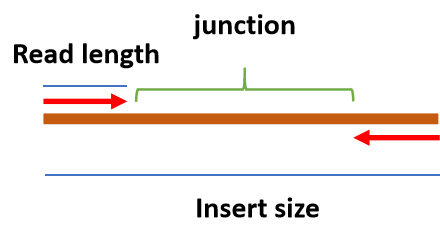
- Illumina测序中的几个名词
- Read length: 测序的DNA片段的碱基长度。
- Insert size: 双端测序时接头(adapter)中间序列的长度。
- Junction: insert序列中间未被测序的部分。
- Flowcell: 是指Illumina测序时,测序反应发生的位置,1个flowcell含有8条lane。
- Lane: 每一个flowcell上都有8条泳道,用于测序反应,可以添加试剂,洗脱等等。
- Raw data: 测序完成后未去接头、引物以及去除低质量序列的数据。
- Clean data: 去除Raw data中的接头序列、linker、低质量reads、长度较短的reads及核糖体RNA和ncRNA产生的reads。
- 数据量: read长度乘以reads数目。
三代测序
三代测序又称为单分子测序,测序过程无需进行PCR扩增,可以产生超长的reads,因此能够跨越高GC含量区域和高度重复区域。目前常用的测序平台包括Pacific Biosciences(PacBio)和Oxford Nanopore。
PacBio以SMRT Cell为载体进行测序反应,SMRT Cell是一张带有纳米孔的超薄金属片。PacBio采用边合成便测序的方式,测序反应在纳米孔中进行,一个纳米孔中固定一个DNA聚合酶和一条DNA模板。延伸反应的过程中检测dNTP荧光信号以确定碱基顺序。
Oxford开发的纳米单分子测序技术属于真正的实时测序,它基于电信号来判读碱基。
| Platform\Instrument | Throughput (Gb) | Read length (bp) | Strength | Weakness |
|---|---|---|---|---|
| Sanger sequencing | ||||
| ABI 3500/3730 | 0.0003 | Up to 1 kb | Read accuracy and length | Cost and throughput |
| Illumina | ||||
| MiniSeq | 1.7–7.5 | 1×75 to ×150 | Low initial investment | Run and read length |
| MiSeq | 0.3–15 | 1×36 to 2×300 | Read length, scalability | Run length |
| NextSeq | 10–120 | 1×75 to 2×150 | Throughput | Run and read length |
| HiSeq (2500) | 10–1000 | ×50 to ×250 | Read accuracy, throughput | High initial investment, run |
| NovaSeq 5000/6000 | 2000–6000 | 2×50 to ×150 | Read accuracy, throughput | High initial investment, run |
| IonTorrent | ||||
| PGM | 0.08–2 | Up to 400 | Read length, speed | Throughput, homopolymers |
| S5 | 0.6–15 | Up to 400 | Read length, speed | Homopolymers |
| Proton | 10–15 | Up to 200 | Speed, throughput | Homopolymers |
| Pacific BioSciences | ||||
| PacBio RSII | 0.5–1 | Up to 60 kb | ead length, speed (Average 10 kb, N50 20 kb) | High error rate and initial |
| Sequel | 5–10 | Up to 60 kb | Read length, speed (Average 10 kb, N50 20 kb) | High error rate |
| Oxford Nanopore | ||||
| MInION | 0.1–1 | Up to 100 kb | Read length, portability | High error rate, run length |
常见序列格式
Fastq
我们得到的下机序列一般为fastq格式,每一条read包含4行,第一行为测序仪器信息以及测序信息,第二行为碱基序列,第三行一般无信息,第四行为对应第二行中每个碱基的测序质量信息。

| Strings | Description |
|---|---|
| @ST-E00310 | The unique instrument name |
| 147 | The run id |
| HVT25CCXX | The flowcell id |
| 3 | Flowcell lane |
| 1011 | The number within the flowcell lane |
| 13382 | ‘x’-coordinate of the cluster within the title |
| 1819 | ‘y’-coordinate of the cluster within the title |
| 1 | The number of a pair, 1 or 2 (paired-end or mate-pair reads only) |
| N | Y if the read fails filter (read is bad), N otherwise |
| 0 | 0 when none of the control bits are on, otherwise it is an even number |
| TGAAGACA | Index sequence |
Fasta
FASTA格式为文本文档,内含核苷酸或氨基酸序列以及其IDs。每条序列包含两部分,第一部分为ID及注释信息,以 “>” 开头,at the start, 第二部分为核苷酸序列或氨基酸序列。

Genbank
GenBank格式包含了基因组序列和注释信息。

GFF3
GFF3 (Generic Feature Format version 3) 格式描述了序列的特征,每一行含有9列数据,列与列之间以制表符分割。
基因组拼接基本概念
测序深度
测序深度(Sequencing depth)指测序得到的总碱基数(read长度x reads数目)与待测基因组大小的比值。假设一个基因组大小为2M,测序深度为10X,那么获得的总数据量为20M。
测序覆盖度
指测序获得的序列占整个基因组的比例。由于基因组中的高GC、重复序列等复杂区域的存在,测序获得的序列经常无法覆盖基因组上所有的区域。例如覆盖度是96%,表明还有4%的序列区域未测到。
Read、Contig、Scaffold
测序得到的序列被称作reads,在一个read中连续的N个碱基所组成的序列称作k-mer,把k-mer集合拼接起来形成的长DNA序列称为contig。通过pair ends信息将contigs按顺序进行排列得到scaffold。
N50
将contigs或scaffolds根据长度从大到小排列并累加,当其累计长度达到全部组装序列总长度的50%时,加上去的最后一个contig或scaffold的大小即为N50的大小,N50是评价基因组拼接质量的重要参数。
参考文献
Besser J, Carleton HA, Gerner-Smidt P, Lindsey RL, Trees E. Next-generation sequencing technologies and their application to the study and control of bacterial infections. Clinical Microbiology and Infection, 2018, 24: 335-341