摘要
多重成像技术能够在单细胞分辨率下同时对组织中的数十种生物分子进行空间分析。从多重组织成像数据中提取生物学相关信息(如细胞表型的空间分布)涉及多项计算任务,包括图像分割、特征提取和空间分辨的单细胞分析。在此,我们提出了一种用于多重组织图像处理和分析的端到端工作流程,该流程整合了之前开发的计算工具,以用户友好和可定制的方式实现这些任务。在数据质量评估方面,我们强调了napari-imc在交互式检查原始成像数据方面的实用性,以及cytomapper R/Bioconductor包在R中进行图像可视化的功能。使用steinbock工具包执行原始数据预处理、图像分割和特征提取。我们展示了两种基于监督像素分类和预训练深度学习模型的细胞分割替代方法。提取的单细胞数据随后在R中进行读取、处理和分析。该方案描述了使用社区建立的数据容器的方法,从而便于应用R/Bioconductor包进行降维、单细胞可视化和表型分析。我们提供了使用imcRtools R/Bioconductor包进行空间分辨单细胞分析的说明,包括群落分析、细胞邻域检测和细胞-细胞相互作用测试。该工作流程之前已应用于成像质谱细胞计数数据,但可以轻松适应其他高度多重成像技术。本方案可由具有基本生物信息学培训的研究人员实施,并且所提供的数据集的分析可在5–6小时内完成。扩展版本可在https://bodenmillergroup.github.io/IMCDataAnalysis/上获得。
介绍
高度多重成像技术能够在组织中同时检测到数十种生物分子(如蛋白质和转录本,也称为“标志物”)。最近建立的多重组织成像技术依赖于荧光标记抗体的循环染色技术[1–3],或使用寡核苷酸标记的[4,5]或金属标记的[6,7]抗体等技术。这些技术的关键优势在于,它们允许对组织空间环境中的单个细胞进行深入分析。因此,这些方法已经能够对肿瘤微环境的空间结构[1,8–10]、用于评估细胞类型和趋化因子空间共定位的核酸和蛋白质丰度[11]、病毒感染细胞的空间生态位[12],以及2019年冠状病毒病(COVID-19)感染[13,14]、1型糖尿病进展[15]和自身免疫性疾病[16]期间的病理特征进行分析。
为了充分利用多重成像数据中包含的信息进行生物学和临床分析,无论生物学问题是什么,都必须执行一系列计算步骤。这些步骤包括:(1)对图像进行目视检查,以初步评估数据质量;(2)生物图像处理与分割,以提取用于下游分析的单细胞特征;(3)单细胞分析,如将单细胞数据投影到低维空间、进行聚类和细胞表型分析,以支持探索性数据分析和量化样本之间的生物学差异[8,15,17];(4)利用从多重成像数据中提取的空间信息来检测细胞表型之间的相互作用或避免作用[18]、表征细胞邻域(CNs)[5,10,19]和空间群落[8],以及识别相似细胞的斑块[11]。存在许多计算工具和方法来执行上述步骤;然而,许多工具缺乏互操作性、可扩展性,并且依赖于复杂的自定义脚本。
在此,我们提出了一种简化且易于扩展的多重图像分析端到端工作流程,该流程支持所有关键步骤,并与现有软件实现互操作性。该工作流程最初是为分析成像质谱细胞计数(IMC)数据而开发的,也适用于其他高度多重成像技术生成的数据。我们专门为可视化原始IMC数据开发的napari图像查看器插件napari-imc可实现交互式图像可视化。steinbock工具包可实现多重图像处理,包括数据提取、图像分割和数据导出。具体而言,我们展示了使用监督像素分类[21,22]和预训练深度学习模型[23]的分割方法,以从多重图像中提取空间分辨的单细胞信息。随后,imcRtools R/Bioconductor包将生成的单细胞数据读入R编程语言。通过使用社区建立的数据容器,该工作流程集成了许多先前开发的R/Bioconductor包,以执行信号溢出校正[24],并允许进行低维可视化、质量控制、细胞表型分析、单细胞聚类和图像可视化[17,25]。最后,我们描述了如何以用户友好的方式使用imcRtools包执行空间群落分析、细胞邻域(CN)和空间上下文(SC)检测、细胞斑块表征和细胞表型相互作用分析。工作流程中使用的所有工具都记录详尽,并且旨在通过互操作性、可重复性和用户友好性分析,为接受过基本生物信息学培训的研究人员提供服务。我们描述了该工作流程,强调了常见陷阱,并讨论了针对各个步骤进行故障排除的方法。
程序概述
本方案描述了分析由高度多重组织成像技术生成的数据的基本步骤。整个流程分为九个阶段:交互式成像数据可视化(步骤1-5)、数据预处理(步骤6)、图像分割(步骤7)、单细胞特征提取(步骤8)、将数据读入R(步骤9-13)、溢出校正(步骤14-21)、质量控制(步骤22-27)、细胞表型分析(步骤28-30)和空间分辨单细胞分析(步骤31-35)。图1给出了该程序的概览,而工作流程的详细描述可在https://bodenmillergroup.github.io/IMCDataAnalysis/上查阅。
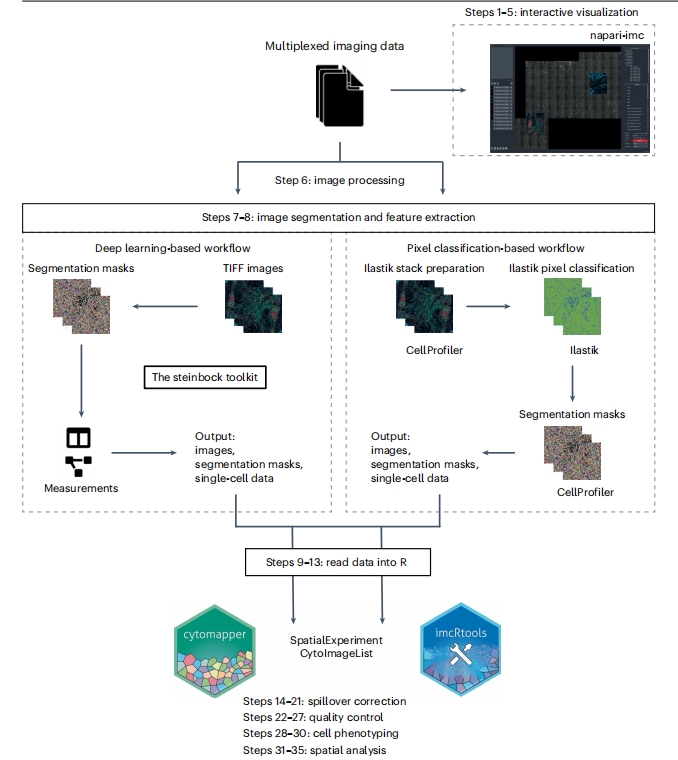
在计算分析之前,通过灵活可视化标记丰度对原始成像数据进行定性检查是手动评估数据质量的关键步骤26。最近开发的多维图像查看器napari能够实现多通道图像的快速交互式可视化20。为了方便检查原始离子束成像(IMC)数据,我们开发了napari-imc插件,该插件使napari能够加载Hyperion成像系统生成的MCD/TXT文件。使用napari-imc,研究人员可以将多重IMC采集数据与仪器获取的光学图像(用于区域选择,即“全景图”)进行叠加,从而便于对原始数据进行定性检查。对于其他多重成像技术,如多重离子束成像(MIBI)7、通过索引共检测(CODEX)5和循环免疫荧光(CyCIF)1,这些技术无法提供生物样本的固有多模态视图(这些数据相当于IMC采集数据,而非IMC全景图),成像数据通常可以转换为napari原生支持的文件格式。
多通道图像处理能够实现多重成像捕获的生物实体的定量分析。单个处理步骤包括图像提取和预处理、图像分割以及生物对象(如细胞)的量化。本文介绍了如何使用Ilastik21和CellProfiler22执行基于随机森林的监督像素分类图像分割(以下简称“基于像素分类的分割”)。这种方法之前曾被用于从多重图像中提取单细胞信息,以进行组织空间表征8,13,15,27。作为一种替代策略,我们还提供了使用深度学习支持的Mesmer分割算法23(以下简称“基于深度学习的分割”)执行基于预训练神经网络的全自动细胞分割的指南。我们的steinbock多通道图像处理工具包提供了基于深度学习的分割方法。
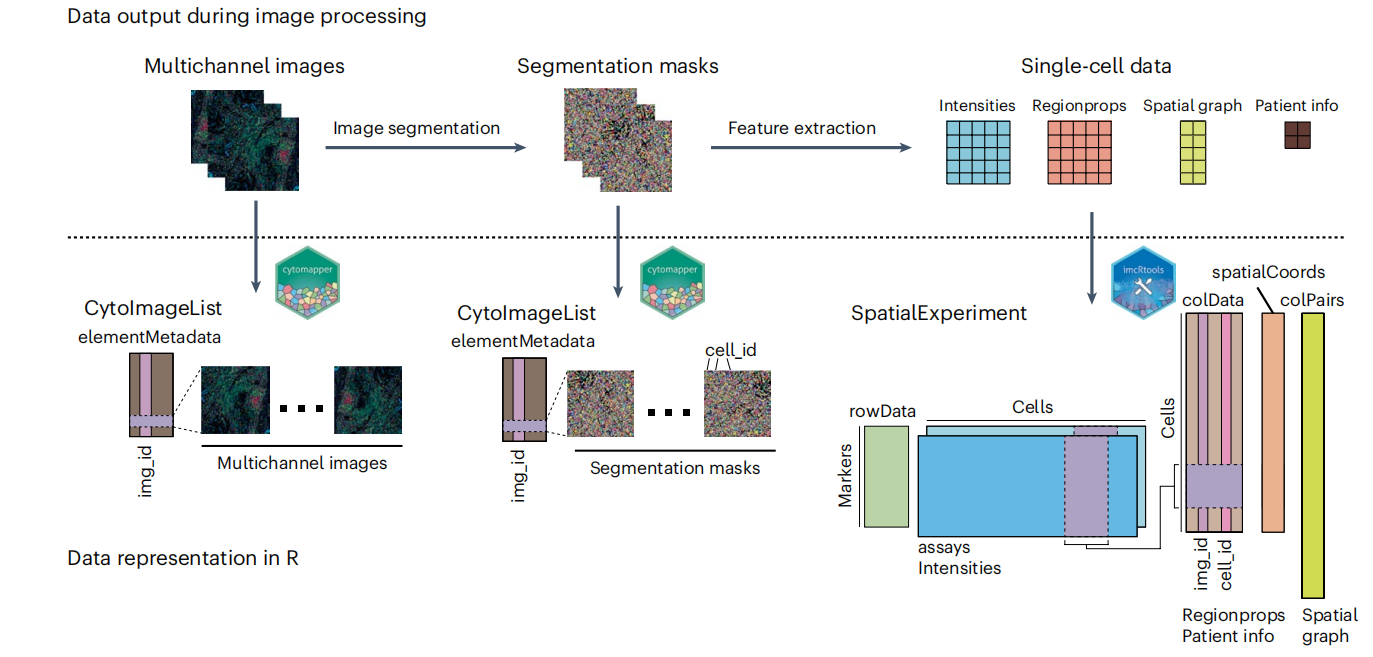
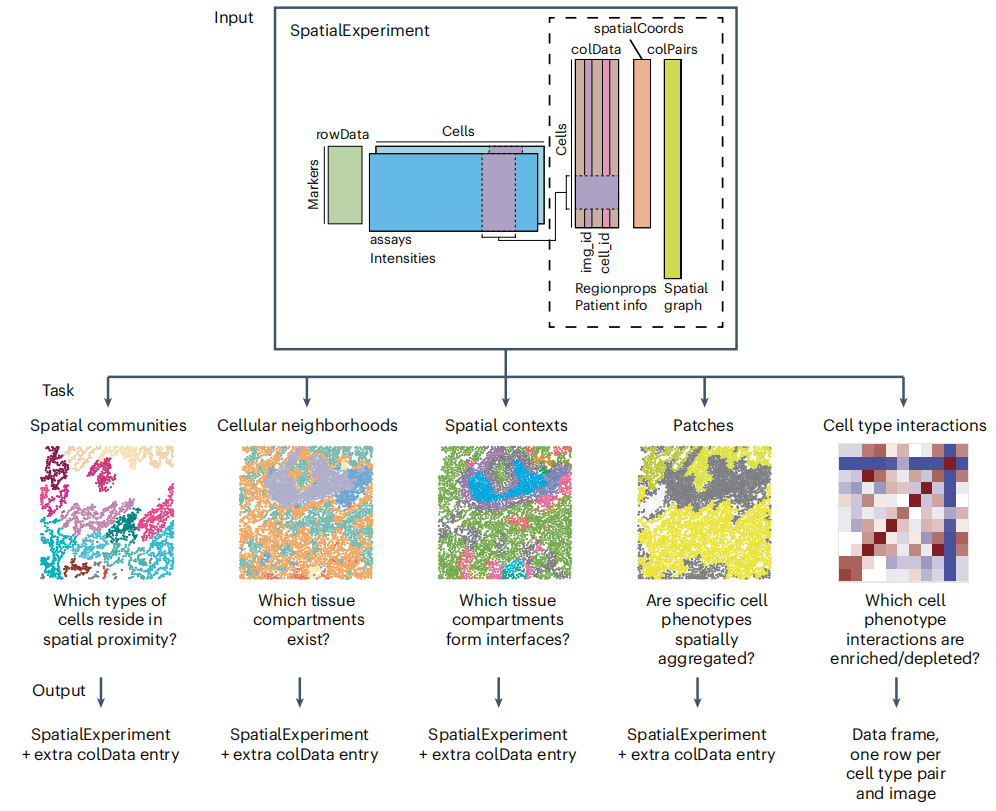
图像分割和空间分辨单细胞数据提取后,将执行下游分析步骤以表征组织环境中的细胞。为此,分别使用imcRtools和cytomapper R/Bioconductor软件包将单细胞数据和图像读入R(图2)。初步数据处理步骤包括溢出校正,以纠正相邻通道间的伪信号24,以及分割和样本质量控制。通过将单细胞数据存储在R/Bioconductor的SpatialExperiment28对象中,该工作流程促进了单细胞数据分析步骤,包括使用现有工具进行聚类、降维、可视化和批次校正17,29,30。为了检测具有相似标记表达(以下简称“细胞表型”)的细胞群,我们描述了两种基于标记真实数据的单细胞聚类和分类的替代方法。最后,我们描述了imcRtools软件包支持的一系列空间数据分析任务,以表征空间组织结构。我们展示了如何检测空间肿瘤和间质群落,这些群落可用于表征不同细胞表型之间的高阶相互作用8。为了结合细胞表型信息检测空间组织结构,我们使用imcRtools软件包将细胞聚合成细胞邻域(CNs)5,10和超级细胞(SCs)19。作为检测空间组织结构的另一种有监督方式,我们重点介绍了一种补丁检测方法,该方法可用于分析预定细胞表型的空间聚集11。最后,为了评估某些类型的细胞表型在图像中的空间相互作用是否增加或减少,我们描述了使用基于置换的测试策略18。
要使用此工作流程,需要掌握R编程语言的基础知识。这包括安装软件包、执行函数、理解函数参数以及如何访问函数文档的能力。此外,研究人员应能够在命令行上执行简单操作,如更改目录。
综上所述,本方案强调了使用最近开发的工具以用户友好和可重复的方式执行端到端的多重图像分析,包括图像可视化和分割、数据质量控制以及单细胞和空间分析。
工作流的应用
工作流的先前应用
之前,所提出的方法已被用于从多重成像数据中提取空间分辨的单细胞特征,以表征组织结构和其与疾病进展的关系8,9,11,15。该方法中描述的图像可视化策略已被用于显示子宫内膜异位症样本的复合图像31,以及在胰岛32和淋巴组织33的分割掩膜上绘制细胞表型和细胞表达的空间图。基于监督像素分类的图像分割方法已被应用于从多重成像数据中提取和分析空间分辨的单细胞数据,以绘制乳腺癌8,9,18,27、1型糖尿病15、COVID-19感染期间的肺和皮肤损伤13,14以及人肾组织空间结构34的特征图。steinbock工具包支持的通过应用预训练的深度学习模型进行图像分割的方法,之前已被用于研究药物筛选期间非小细胞肺癌球体中的标记表达35,以及表征转移性黑色素瘤中的趋化因子环境11。在本文方法中,我们还描述了一种细胞表型分析策略,即根据图像中细胞的标记表达进行手动标记,然后用于分类。该策略之前已被用于识别转移性黑色素瘤11和COVID-19患者皮肤样本14中的细胞表型。在方法的最后部分,我们提供了空间单细胞分析策略的指导。空间群落检测方法之前已被用于表征乳腺癌的空间结构及其与单细胞病理学组的关系8。细胞邻域(CN)和超级细胞(SC)检测已被用于了解结直肠癌样本内空间域的作用和组装10,19。本文描述的补丁检测方法已被用于识别趋化因子环境的细胞成分11。最后,用于识别细胞表型之间相互作用或避免作用的测试策略,之前已被用于表征乳腺癌8,18,27、转移性黑色素瘤11和胰腺组织15中的局部细胞表型相互作用。
对于图像分割,我们描述了两种基于监督像素分类和预训练深度学习模型的替代方法。基于监督像素分类的分割方法通过调整CellProfiler管道提供了灵活性,并可以通过训练特定的Ilastik分类器15扩展到分割更大的组织结构,如胰岛。该管道之前已在多项研究中用于从多重图像中提取单细胞信息8,9,13–15,18,27。然而,最初开发的基于像素分类的分割管道需要自定义脚本、手动软件安装,并且仅通过使用随机森林的监督像素分类提供图像分割策略。为了促进多通道图像处理,我们因此开发了steinbock工具包,该工具包可实现数据提取、图像分割并且以用户友好的方式实现数据导出。它还通过使用启用深度学习的Mesmer分割算法,基于预训练的神经网络提供全自动细胞分割。steinbock工具包易于使用,旨在通过提供版本化和容器化的环境,确保在不同运行和平台之间的可重复性。
工作流的技术中立应用
所提出的方法之前已被用于IMC(多标记质谱流式细胞术)生成的数据的端到端分析。然而,除了个别明确指示且专门为IMC定制的步骤外,所提供的工具还可应用于其他多重成像技术生成的数据。但是,一些迭代式多重成像技术需要额外的预处理步骤,如图像拼接和配准1,5,而这些步骤并未包含在所提出的工作流中。为了使本文描述的工具适用于给定的多重组织成像数据集,图像必须作为(拼接和共配准的)多通道TIFF文件提供。
此外,如果使用了免疫荧光通道来检测细胞核和细胞膜36,则所描述的工具还可用于分析空间转录组数据,如seqFISH。对于此类数据,steinbock工具包可用于图像分割和单细胞数据提取,imcRtools包可执行空间数据分析,而cytomapper包则允许在分割掩膜上可视化细胞表型。
工作流的扩展
所提出的方法提供了执行多重成像数据基本分析所需的所有工具。然而,根据研究问题,可以使用额外的工具和软件包来扩展所提出的方法。对于查看多通道图像,QuPath26提供了灵活且用户友好的可视化选项,这些选项可在TIFF图像上操作。steinbock工具包允许以流式细胞术标准(FCS)文件的形式导出单细胞数据,以便与FCS Express或Cytobank一起使用。此外,由于可以将单细胞数据以anndata对象37的形式导出,因此可以通过Python中的单细胞和空间数据分析来扩展该方法。可以使用slingshot38进行轨迹分析,spicyR支持检测细胞类型共定位变化32,而edgeR17,39可用于执行差异丰度测试。有关单细胞分析方法的全面概述,请参阅《使用Bioconductor编排单细胞分析》一书,该书可在https://bioconductor.org/books/release/OSCA/上获取。
与其他方法的比较
图像查看器
为了交互式多通道图像可视化,之前已开发了包括histoCAT18、CytoMap40、cytokit41和vitessce42在内的多个图像查看器。然而,现有的图像查看器不支持读取存储在专有数据格式中的原始IMC数据所包含的全部信息。本文描述了使用napari-imc专门读取原始IMC数据,并将多重IMC采集叠加到全景图上,从而能够在组织上下文中检查图像。
图像分割方法
对于图像分割,之前应用的两种基于像素分类和深度学习模型的方法已被纳入工作流。基于像素分类的分割方法依赖于使用Ilastik进行像素分类和使用CellProfiler进行细胞分割。该分割方法的简化版本已在imcyto管道43中实现。近年来,已开发了诸如StarDist44、Cellpose45和Mesmer23等启用深度学习的图像分割方法,以在多种组织类型和成像平台上实现人类水平的分割性能23。这些方法通常需要自定义脚本和特定的数据预处理步骤。我们已将启用深度学习的Mesmer分割算法纳入steinbock,因为它已针对通过多种多重成像技术获取的数据进行了专门训练。
单细胞与空间分析工具
单细胞与空间数据分析依赖于一系列交互式和程序化工具的支持。专为某些多重成像平台设计的图形用户界面(GUI)软件操作简便,可实现多通道图像和单细胞数据的联合可视化18,40,41,46。然而,这些工具往往与其他框架的互操作性较差,且难以扩展到其开发数据类型之外的其他用例。本文介绍了一些用户友好的计算工具,能够以简化的方式执行所有关键分析步骤,并确保与现有软件包(如Bioconductor项目中的软件包47)的互操作性。对于程序化的单细胞与空间数据分析,最近开发了squidpy软件包48用于在Python中分析数据,而giotto49则在R中进行类似的分析。这两个软件包都提供了大量用于空间分辨单细胞分析的功能,steinbock工具包则允许导出数据以便与squidpy结合使用。相比之下,Bioconductor项目47中的imcRtools和cytomapper软件包用于空间数据分析和图像可视化,因此能够与使用标准化数据类的众多单细胞数据分析方法相结合17,28。
分析流程
CellProfiler22、Ilastik21、QuPATH26和cytokit41等单个工具,以及包含imcyto43、MIRIAM50和mplexable51的工作流,可执行图像处理和分割。这些工具和工作流侧重于多重图像分析的特定处理步骤,通常与空间分辨单细胞分析等下游分析任务缺乏互操作性。最近,开发了一个名为MCMICRO的全面流程,用于从使用各种成像技术获取的多通道图像中提取和分析单细胞数据52。MCMICRO流程允许执行迭代染色方法通常所需的图像处理步骤,包括图像拼接和配准、图像分割和特征提取。对于单细胞和空间数据分析,该流程提供了使用phenograph53进行单细胞聚类和使用CN检测54的模块,并且可以通过开发新模块来扩展该流程。本文所述的方案提供了超越MCMICRO流程中模块的空间分辨单细胞分析策略。具体而言,在简化图像分割、特征提取并将数据读入R之后,本文重点介绍了常用的单细胞分析工具,以执行降维、单细胞可视化、批次校正和聚类以及基于分类的表型分析。此外,通过使用SpatialExperiment容器来存储空间分辨单细胞数据,该工作流可与许多当前和未来开发的用于空间和单细胞数据分析的Bioconductor软件包相结合。
数据生成的实验设计
在应用本文所述的方案分析多重成像技术生成的数据之前,研究人员必须仔细考虑要成像的样本数量、测量的感兴趣区域的数量和位置、样本质量以及抗体鸡尾酒的特异性和敏感性55。样本大小和感兴趣区域(ROI)大小主要受研究性质和材料可用性的影响。建议进行预实验,以估算最佳采集数量和大小,从而描绘出感兴趣的组织特征56,57。
在最终数据采集之前,需要针对感兴趣的组织类型和包埋策略(即福尔马林固定石蜡包埋(FFPE)或最佳切割温度复合物(OCT))专门评估每种抗体的染色质量。为了评估技术差异,如染色效率随时间的变化或实验变化,建议与实验样本平行地对控制样本(如细胞系团块)进行染色和采集。这些控制细胞团块也应使用与主要样本相同的流程进行分析,以评估技术差异。
本文所述的数据分析方案假设所有样本都是使用相同的抗体面板采集的。如果使用不同的面板,则应对每个子批次样本分别应用该方案。下文讨论了评估数据质量的几种方法,并提出了在分析步骤中纠正和/或避免技术偏差的方法。
局限性
多通道图像处理的内存和存储限制
基于像素分类的分割方法在处理大量图像时会遇到内存和运行时问题。首先,在Ilastik中加载和标记许多大文件会消耗大量内存并减慢像素标注的速度。我们通过裁剪图像来解决这个问题,并需要决定是加载较小的裁剪图像还是较少的图像到Ilastik中。此外,通过启用Ilastik中的“实时模式”来即时重新训练分类器也会消耗大量内存。这个问题可以通过关闭“实时模式”进行标注来解决。最后,所提供的CellProfiler流程会从图像中提取所有可用特征。对于大型数据集,这可能会导致生成存储单细胞特征的大型(多个千兆字节)CSV文件。因此,我们建`议手动选择对下游分析感兴趣的特征。
与其他工具的兼容性
steinbock工具包主要处理存储在未压缩TIFF文件中的图像,这需要足够的计算资源。在使用steinbock时,可以通过压缩原始数据进行长期存储并启用图形处理单元(GPU)支持图像分割来减轻与数据集和/或图像大小相关的问题。通过批量处理图像瓦片而不是完整图像来支持大型全玻片图像(大小为数十GB)的处理。此外,steinbock尽可能访问磁盘上的TIFF文件,而不是将完整图像加载到内存中。在使用深度学习支持的Mesmer算法进行全自动图像分割时,底层预训练神经网络可能并非对所有数据集都能立即表现出色。steinbock工具包可使用–preprocess/–postprocess命令行选项来微调算法参数(参见“故障排除”部分)。或者,也可以采用基于像素分类的监督图像分割方法,其中用于分割的像素分类器可以根据当前数据集进行定制(重新训练)。
R中的图像可视化
cytomapper包目前支持以HDF5格式在磁盘上存储图像,这有助于处理大型图像。然而,在R的原生图形设备中一次性显示大型全玻片图像计算成本高昂,我们建议先对图像进行分块,然后并排可视化单个图像块。
选择正确的聚类数量
我们在两种情况下描述了聚类方法:首先,是识别细胞表型(第28步),其次,是检测拷贝数(CN)(第32步)。在这两种情况下,选择聚类方法和聚类数量都不简单,需要测试不同的参数,直到忠实地概括所研究系统的先前生物学知识或估计聚类稳定性(例如,一些聚类在不同的参数设置下不会发生变化)。我们在第28步中讨论了合适的参数扫描。然而,为了检测CN,如果可能的话,最好对要检测的组织结构有预先的预期,并相应地调整聚类参数。对于无监督的CN检测,可以测试多种聚类参数设置和方法来估计结果的稳健性。
随着细胞数量增加的可扩展性
本协议中介绍的大多数函数在分析空间解析的单细胞数据时都可以处理数百万个细胞。然而,在进行非线性降维、聚类(除k均值聚类外)、图像块检测(当扩展许多图像块时)和细胞表型相互作用测试时,可能会出现内存和运行时问题。这些问题可以通过imcRtools中实现的函数来缓解,这些函数易于并行化或使用近似方法来检测最近邻。对于相互作用测试函数,当同时分析数百万个细胞时,可能会出现内存问题。作为一种解决方法,可以按图像子集处理数据集,因为统计信息是按图像计算的。
材料
设备
硬件
• 要运行该工作流程,需要一台装有最新版本的Windows、Mac或Linux操作系统(OS)的电脑。随着数据集大小的增加,所需的内存也会增加,我们建议使用至少8GB的随机存取存储器(RAM)来分析所提供的数据集(14600×600像素的图像,40个通道,47859个细胞)。对于更大的数据集(数百万个细胞),我们建议使用至少64GB的RAM。或者,如果可以安装Docker(见“设备安装”部分),则可以使用高性能电脑。对于本手稿,工作流程是在MacOS Big Sur(11.7.4)、2.7 GHz四核Intel Core i7、16 GB 2133 MHz LPDDR3上运行的。
软件
• napari & napari-imc(针对IMC):使用多维图像查看器napari(https://napari.org)以及用于加载IMC文件的napari-imc插件(https://github.com/BodenmillerGroup/napari-imc)来可视化和检查原始的多重成像数据。我们在一个新的conda(https://conda.io)环境中安装了Python 3.9.12(https://www.python.org)、napari 0.4.16和napari-imc 0.6.5;安装说明见下文。
• 基于深度学习的分割:使用多通道图像处理工具包steinbock(https://bodenmillergroup.github.io/steinbock)对多重成像数据进行预处理,执行图像分割并提取单细胞数据。我们使用Docker Desktop 4.9.0 for Mac从GitHub容器注册表中拉取了steinbock Docker容器v0.16.0;安装说明见“设备安装”部分。
• 基于像素分类的分割:使用基于随机森林的像素分类和基于分水岭的细胞分割方法进行多通道图像处理,使用的是基于Ilastik/CellProfiler的分割管道v3.6(https://bodenmillergroup.github.io/ImcSegmentationPipeline/);安装说明见“设备安装”部分。此外,要使用该管道,还需要安装以下软件:
– Ilastik:在细胞分割之前用于像素分类的Ilastik软件21,可从https://www.ilastik.org/download.html安装。本工作流程使用的版本是v1.4.0。
– CellProfiler:用于分割单个细胞的CellProfiler软件22,可在Windows(64位)和MacOS(10.14+)上从https://cellprofiler.org/previous-releases安装。本工作流程使用的版本是v4.2.1。
• R设置:图像处理后的下游分析使用的是统计编程语言R,可从https://cran.r-project.org/按照特定操作系统的说明进行安装。本工作流程使用的版本是v4.3.0。
RStudio软件为R中的数据分析提供了一个易于使用的图形用户界面(GUI)。它可以从https://www.rstudio.com/products/rstudio/download/安装。为了执行所展示的工作流程,我们使用了RStudio Server版本2023.03.0。
以下是本工作流程中使用的主要R库的版本。此外,补充说明1列出了工作流程中使用的所有R库的软件版本。
SpatialExperiment版本1.10.0(参考文献28)
SingleCellExperiment 1.22.0(参考文献17)
CATALYST版本1.24.0(参考文献24)
imcRtools版本1.6.0
scuttle版本1.10.0(参考文献58)
scater版本1.28.0(参考文献58)
batchelor版本1.16.0(参考文献30)
bluster版本1.10.0
scran版本1.28.0(参考文献59)
caret版本6.0-94
Cytomapper 版本 1.12.0
DittoSeq 版本 1.12.0(参考文献29)
Tidyverse 版本 2.0
数据
IMC 示例数据:我们提供了一个小型IMC数据集作为示例,以展示多通道图像处理和空间分辨单细胞数据分析。这些数据是使用Hyperion成像系统(Standard BioTools)在大型适应性癌症患者队列的综合免疫分析(IMMUcan)项目中生成的。数据可访问于https://zenodo.org/record/7575859,包含以下文件:
Patient1.zip, Patient2.zip, Patient3.zip, Patient4.zip:四个患者样本的原始IMC数据。每个ZIP归档包含一个文件夹,其中包含一个MCD文件(所有采集的数据和元数据)和多个TXT文件(每个采集一个文件,无元数据)。
compensation.zip:此ZIP归档包含一个文件夹,其中包含一个MCD文件和多个TXT文件。采集了“溢出载玻片”的多个点,每个TXT文件根据点样金属命名。这些数据用于通道溢出校正(步骤14–21)。
sample_metadata.csv:此文件将每个患者与其癌症类型相关联(SCCHN,头颈癌;BCC,乳腺癌;NSCLC,肺癌;CRC,结直肠癌)。
我们还提供了steinbock格式的panel.csv文件,其中包含实验中使用的每个抗体或通道的元数据。在此文件中,“channel”列指示用于标记每个抗体的金属同位素。“name”列包含每个抗体的蛋白质靶点。“keep”列指示哪些通道是从原始数据中提取的,因此在下游分析中保留。重要的是,“keep”列设置为1的行的顺序与提取的多通道图像中通道的顺序相匹配。最后,“deepcell”列指示在使用DeepCell/Mesmer进行自动图像分割时使用了哪些通道(即聚合):核通道设置为1,细胞质通道设置为2,其他/未使用的通道留空。该文件可访问于https://zenodo.org/record/7624451。
此外,我们还提供了包含先前筛选细胞的SpatialExperiment对象。这些细胞构成了基于分类的表型分析中的真实细胞表型标签(步骤28)。数据可访问于https://zenodo.org/record/7647079。
设备设置
安装说明
除非另有说明,否则本节中的命令必须在命令行上执行(Windows上的“命令提示符”或“Anaconda提示符”,Linux/Mac OS上的“终端”)。
- napari 和 napari-imc:
根据https://docs.conda.io/projects/conda/en/latest/user-guide/install/上的说明安装conda包管理器。
创建一个新的conda环境,包含Python 3.9:
1 | conda create -n napari-imc -y python=3.9 |
- 激活conda环境并安装napari和napari-imc:
1 | conda activate napari-imc |
基于深度学习的分割:有关安装容器化steinbock工具包的说明,可在steinbock在线文档中找到(https://bodenmillergroup.github.io/steinbock/)。特别是,要运行steinbock容器,需要先安装Docker(请参阅在线说明)。对于本手稿,我们使用以下别名运行steinbock:
1
alias steinbock="docker run -v /path/to/data/steinbock:/data -u (id−u):(id -g) ghcr.io/bodenmillergroup/steinbock:0.16.0"
▲ 关键说明:在上述命令中,需要将/path/to/data/steinbock替换为预期的steinbock数据/工作目录。
- 基于像素分类的分割:基于像素分类的分割流程的预处理步骤是使用自定义脚本在Python中执行的。要设置预处理脚本,需要执行以下步骤:
1 | git clone --recursive https://github.com/BodenmillerGroup/ImcSegmentationPipeline.git |
- 创建imcsegpipe的conda环境:
1 | cd ImcSegmentationPipeline |
- 通过打开CellProfler的图形用户界面(GUI),选择Preferences并将CellProfler插件目录设置为path/to/ImcSegmentationPipeline/resources/ImcPluginsCP/plugins,来配置CellProfler以使用所需的插件,然后重启CellProfler。
▲ 关键说明:将此路径调整为下载的CellProfler插件所在的位置。按照上述方式克隆仓库时,插件位于resources/ImcPluginsCP/plugins中。
R库:该工作流程强调了使用cytomapper、imcRtools和其他R包。以下R命令会安装所有相关的R包:
1
2
3
4
5
6
7
8
9
10if (!requireNamespace("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
BiocManager::install(c("pheatmap", "viridis",
"tiff", "distill", "ggrepel", "patchwork",
"mclust", "RColorBrewer", "uwot", "Rtsne", "caret",
"randomForest", "ggridges", "gridGraphics", "scales",
"CATALYST", "scuttle", "scater", "dittoSeq",
"tidyverse", "batchelor", "bluster","scran",
"cytomapper", "imcRtools"))
可以从GitHub或Bioconductor直接安装R包的开发版本,同时指定开发版本。开发版本通常包含发行版中尚未提供的功能;然而,对于使用描述的工作流程,无需从开发源安装R包。
为确保工作流程的可重复性,我们还提供了一个Docker容器,该容器打包了所有相关的R库。以下命令行调用可获取Docker镜像:
1 | docker pull ghcr.io/bodenmillergroup/imcdataanalysis:2023-05-01 |
使用Docker容器重现分析的说明可在https://github.com/BodenmillerGroup/IMCDataAnalysis/tree/main/publication上查看。此工作流程中显示的分析是在Docker容器内执行的;但是,R包也可以按照上述说明手动安装。
数据下载
在R语言中,可以按照以下步骤下载IMC示例数据和门控细胞:
1 | options(timeout = 10000) # 设置超时时间为10000秒 |
▲ 关键说明:下载的数据和相关的面板文件用于steinbock。若要下载用于基于像素分类的分割流程的示例数据,请遵循步骤6B(iii)。
流程
原始成像数据检查(IMC专用)
● 耗时:20–40分钟
除非另有说明,否则本节中的命令必须在命令行中执行(在Windows上为“命令提示符”或“Anaconda提示符”,在Linux/Mac OS上为“终端”)。
- 激活之前创建的napari-imc conda环境并启动napari:
1 | conda activate napari-imc |
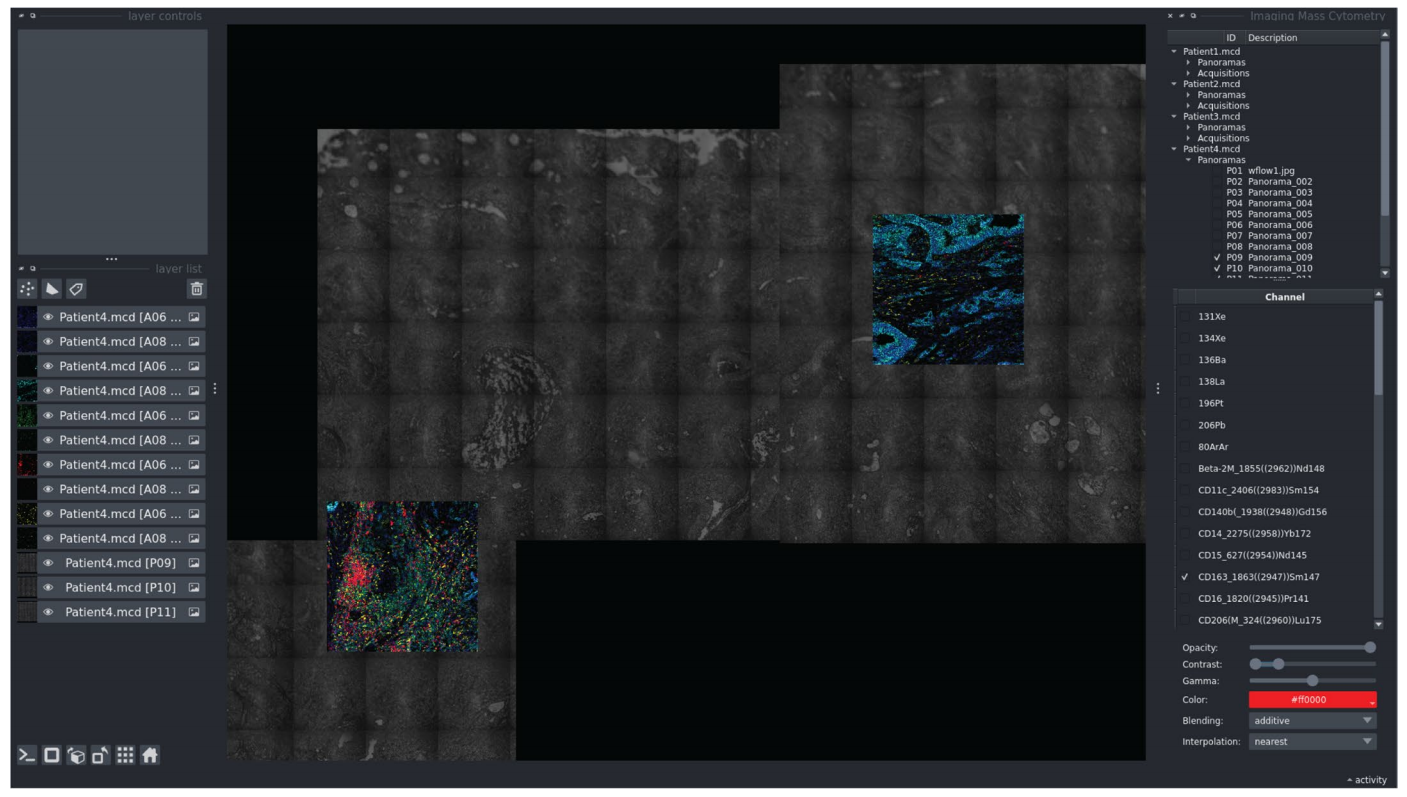
在napari中,使用“文件”→“打开文件(s)”打开包含原始IMC数据的MCD文件。打开文件后,将出现napari-imc小部件(图3,右侧)。
在MCD文件树(napari-imc小部件,顶部)中,选择要在napari主面板中以叠加方式可视化的全景图和/或采集数据。
对于上一步中选择的采集数据,选择要可视化的通道(napari-imc小部件,中部),并调整通道属性,如颜色和对比度(napari-imc小部件,底部)。
使用napari中的平移/缩放功能对合成图像进行定性检查。
多重组织图像处理
除非另有说明,否则本节中的命令必须在命令行中执行(在Windows上为“命令提示符”或“Anaconda提示符”,在Linux/Mac OS上为“终端”)。
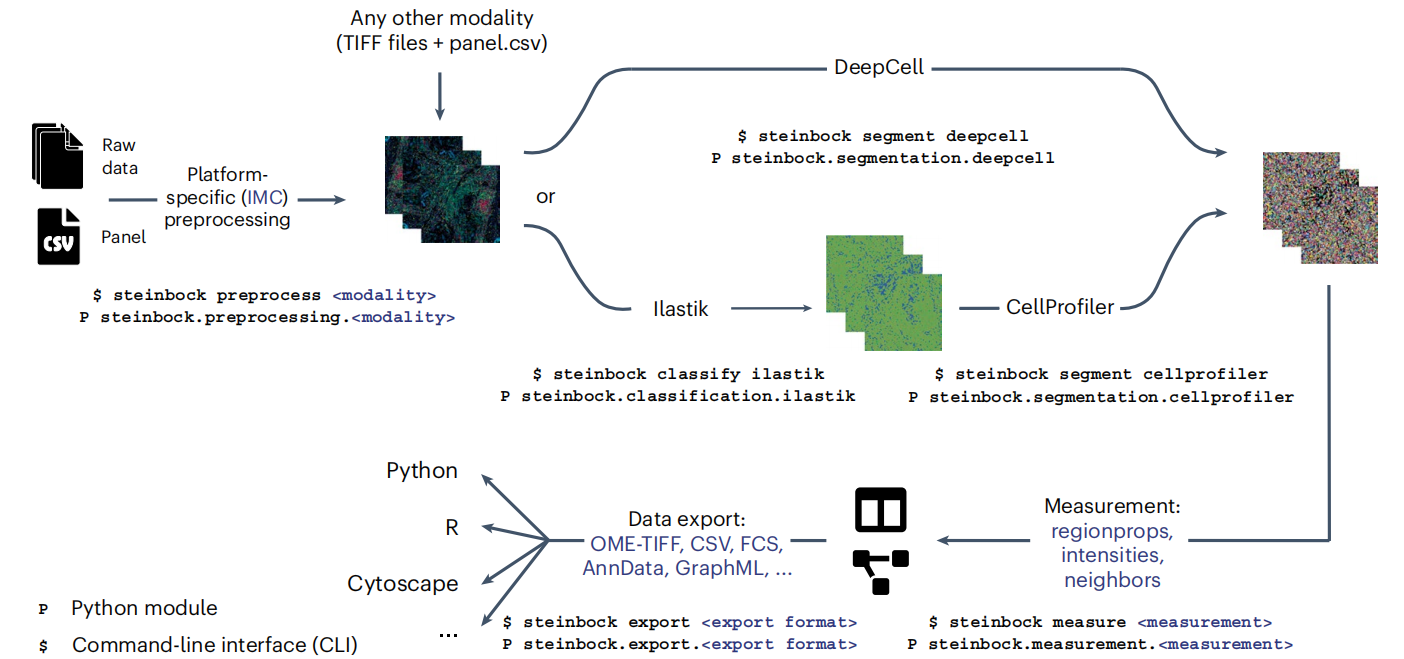
多重组织图像处理通常包括图像提取和预处理、图像分割以及单细胞数据提取等步骤。在此,我们提出了两种用于分割多重组织成像数据的替代工作流程:
(A)基于steinbock的深度学习工作流程,steinbock是我们用于多重组织图像处理的容器化工具包(图4,DeepCell变体)。该工作流程可通过命令行界面进行,并利用DeepCell包中实现的深度学习Mesmer算法,基于预训练神经网络以全自动方式分割细胞。所有steinbock命令的输入和输出文件及目录均可通过命令行选项进行控制。若未明确指定这些选项,steinbock命令行界面将预期以下数据/工作目录结构:
steinbock数据/工作目录
├── raw(用户提供,从原始数据开始)
├── panel.csv(用户提供或从原始数据生成,步骤6(A))
├── img(用户提供,从TIFF文件开始,或从原始数据提取,步骤6(A))
├── masks(步骤7(A)生成,细胞分割掩码)
├── intensities(步骤8(A)生成,平均细胞像素强度)
├── regionprops(步骤8(A)生成,形态细胞特征)
├── neighbors(步骤8(A)生成,空间细胞邻域列表)
(B)使用Jupyter笔记本和CellProfiler管道简化的基于像素分类的工作流程(图5)。在此工作流程中,首先使用Ilastik软件手动训练一个针对当前数据集定制的随机森林像素分类器。然后,使用CellProfiler管道基于像素分类来分割单个细胞。
基于像素分类的分割管道的文件夹结构如下所示。该管道期望将原始IMC数据放置在“raw”文件夹中,或将OME–TIFF数据放置在“ometiff”文件夹中。
基于Ilastik/CellProfiler的分割管道数据/工作目录
├── raw(用户提供,当从原始数据开始时)
├── analysis
├── cpin
├── ometiff(用户提供,当从OME-TIFF开始时,或在步骤6(B)中从原始数据生成)
├── cpout(在步骤6-8(B)中生成;最终输出文件)
├── histocat(在步骤6(B)中生成;用于histoCAT导入的文件)
├── crops(在步骤7(B)中生成;Ilastik裁剪图像)
├── ilastik(在步骤6-7(B)中生成;与Ilastik相关的文件)
├── scripts(原始数据处理脚本)
├── resources(CellProfiler管道和插件)
▲ 重要提示:steinbock工具包(图4)也支持此处所示的基于像素分类的分割方法(图5)。为简洁起见,在本方案中,仅展示了steinbock的深度学习工作流程;如需更多信息,请参阅steinbock的在线文档。
这两个工作流程都接受用户提供的多通道图像作为输入,这些图像可以是TIFF文件(steinbock;需放置在“img”文件夹中),也可以是OME–TIFF格式(基于像素分类的分割管道;需放置在“analysis/ometiff”文件夹中)。另外,也可以直接将原始IMC数据提供给这两个工作流程(需放置在“raw”文件夹中,适用于两个工作流程),并使用我们自主研发的readimc Python包(框1)将其自动转换为相应的文件格式。此外,这两种工作流程都需要一个CSV格式的面板文件,用于指定实验中使用的抗体以及在后续处理中应保留的任何额外通道(例如,用于复染的金属61)。这些面板文件可以自动从原始数据中生成(steinbock),也可以由用户手动指定(steinbock和基于像素分类的工作流程)。
框1
原始IMC数据
Hyperion成像系统生成专有MCD/TXT格式的文件,这些文件存储在以下文件夹结构中:
├── {XYZ}_ROI_001_1.txt
├── {XYZ}_ROI_002_2.txt
├── {XYZ}_ROI_003_3.txt
├── {XYZ}.mcd
在这里,{XYZ}定义了文件名,ROI_001、ROI_002、ROI_003是用户为所选ROI定义的名称(描述),而1、2、3表示唯一的采集标识符。在选择ROI时,可以在Standard BioTools软件中指定ROI描述条目。MCD文件包含所有采集到的ROI的原始成像数据和完整元数据,而每个TXT文件则包含单个ROI的数据(不包含元数据)。为了遵循一致的命名方案并捆绑所有元数据,我们建议将文件夹压缩为ZIP文件。每个ZIP文件应仅包含一个MCD文件及其对应的TXT文件,并且ZIP文件的名称应与MCD文件的名称相匹配。
数据预处理(特定于技术)
● 选项A耗时5分钟,选项B耗时10–20分钟
在本方案中,数据预处理是指从原始成像数据中提取多通道图像,并为后续处理做准备。所需步骤取决于成像技术;在此,我们展示了原始IMC数据的预处理,其中包括热点像素过滤步骤(框2)。在图像分割之前,可以进行额外的模态特异性预处理步骤,如IMC去噪62或通道间溢出校正(步骤14–21),以促进后续的图像分析步骤。以前,对于IMC数据,热点像素过滤就足以进行有效的分割。
对于除IMC之外的多重组织成像模态,可以使用steinbock的预处理外部命令将外部预处理过的二维多通道图像转换为steinbock兼容的TIFF文件。例如,迭代多重组织成像方法的外部预处理通常包括诸如拼接和注册对应于单个成像周期的图像等任务63。有关更多详细信息,请参阅steinbock的在线文档。
预处理多通道图像。
(A)基于深度学习的工作流程
(i)在steinbock面板文件格式(表1;请参阅steinbock的在线文档)中手动创建一个panel.csv文件,并将其放置在steinbock的数据/工作目录中。本方案中使用的完整steinbock面板文件可从https://zenodo.org/record/7624451下载。▲ 关键信息:在使用steinbock处理自定义IMC数据集时,可以使用steinbock的预处理imc面板命令(请参阅steinbock的在线文档),从放置在原始数据目录中的MCD/TXT文件中自动生成模板面板文件。此命令将自动填充“通道”和“名称”列,并可作为创建有效面板文件的起点。
(ii)从MCD/TXT文件中提取单个IMC采集,仅保留面板文件中“保留”列设置为1的通道。使用50的阈值过滤热点像素,并将生成的图像存储为TIFF文件。提取图像中通道的顺序由面板文件中通道的顺序(“通道”列)决定。1
steinbock preprocess imc images --hpf 50
◆ 故障排除
(B)基于像素分类的工作流程
(i)创建一个包含列条目“金属标签”、“full”和“ilastik”的面板文件(表2)。一个可用于所提供数据的示例面板文件可在https://zenodo.org/record/7575859上访问。
(ii)在ImcSegmentationPipeline文件夹内,激活之前创建的imc-segpipe conda环境(参见“安装说明”),并启动jupyter-lab:
1
2
conda activate imcsegpipe
jupyter lab
(iii)按照位于“scripts/imc_preprocessing.ipynb”的脚本执行以下预处理任务:
• 根据“scripts/download_examples.ipynb”脚本下载示例数据
• 将文件类型从MCD转换为OME–TIFF
• (可选)将OME–TIFF文件转换为单通道TIFF文件,以便在histoCAT18中使用
• 将OME–TIFF文件转换为热点像素过滤后的多通道TIFF堆栈,用于(1)图像分析(包含在“full”列中的通道)和(2)ilastik像素分类(指定在“ilastik”列中的通道)
框2
热点像素过滤
热点像素过滤的工作原理是将每个像素与其八邻域(即切比雪夫距离为1的相邻像素)进行比较。如果某个像素与其八个相邻像素中的最大值之间的差异超过热点像素过滤阈值,则将该像素设置为最大相邻像素值(“热点像素过滤后”)。在这里,使用的热点像素过滤阈值为50;但是,该阈值可以根据具体的数据集进行调整。
表1 | steinbock面板文件格式中的示例面板文件(摘录)
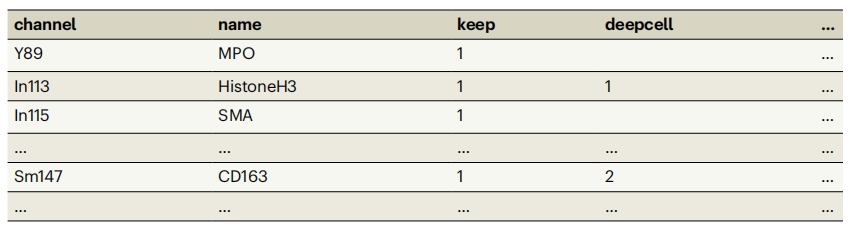
每一行对应于多重成像数据中的一个通道。在IMC(多重同位素标记质量细胞成像)数据中,“通道”列指示用于标记每种抗体的独特金属同位素,该列用于将面板文件和原始数据相关联。“名称”列包含唯一、用户定义的通道名称(例如,每种抗体的蛋白质靶点),这些名称将在派生的单细胞数据和下游分析中使用。“保留”列指示应从原始数据中提取并因此保留以供下游分析哪些通道。重要的是,保留列设置为1的行的顺序将与提取的多通道图像中通道的顺序相匹配。最后,“deepcell”列指示应使用(即聚合)哪些通道,以便使用DeepCell/Mesmer进行全自动图像分割(核通道标记为1,细胞质通道标记为2,其他/未使用的通道该列留空)。本方案中使用的完整steinbock面板文件已从“数据下载”部分的https://zenodo.org/record/7624451下载。
表2 | 基于像素分类的工作流的面板规范

“金属标签”列存储每个采集通道的金属同位素。“全部”和“ilastik”列设置为0或1。“全部”列中的1指定应写入图像堆栈的通道,该图像堆栈稍后将用于提取特征(也称为“全部堆栈”)。“ilastik”列中的1表示将用于Ilastik像素分类的通道,因此将用于图像分割(也称为“ilastik堆栈”)。
图像分割
● 选项A耗时10–20分钟,选项B耗时2–3小时
- 执行图像分割以检测单个细胞,生成并存储为TIFF文件的细胞掩膜。细胞掩膜是与相应输入图像大小相同的单通道灰度图像,其中对应于同一细胞的像素显示相同的灰度值(背景像素的像素值为0)。
(A)基于深度学习的工作流
(i)使用DeepCell软件包中实现的启用深度学习的Mesmer算法,基于预训练神经网络执行全自动细胞分割。在以下命令中,将根据面板文件中的“deepcell”列对通道进行最小-最大归一化和均值聚合。
1
steinbock segment deepcell --minmax
◆ 故障排除
(B)基于像素分类的工作流
(i)为Ilastik像素分类准备图像。
第一步,使用CellProfiler中的“resources/pipelines/1_prepare_ilastik.cppipe”管道为像素标记和分类准备图像。在CellProfiler中导入管道后,首先执行以下步骤:
• 将“analysis/ilastik”文件夹拖放到“Images”下的窗口中
• 在“输出设置”中,将“analysis/crops”文件夹设置为“默认输出文件夹”
管道将读取“analysis/ilastik”文件夹中所有包含“_ilastik”后缀的多通道图像。以下步骤是管道的一部分:
• 计算所有通道的平均强度,乘以100以匹配实际标记的范围,并将其作为第一通道添加到ilastik堆栈中
• ilastik堆栈的像素按两倍比例放大。这将使图像中的像素数量增加四倍,并便于像素标记
▲ 关键提示:为便于视觉像素标记,在使用如IMC等分辨率较低的复用成像技术时,我们建议放大像素。对于基于荧光的技术,可以跳过此步骤。
• 将ilastik堆栈裁剪成较小的视野。默认情况下,这些是500×500像素的裁剪图像;对于大型数据集,较小的图像裁剪即可满足需求
• 将放大的ilastik堆栈以HDF5格式(文件名以_s2.h5结尾)保存到“analysis/ilastik”文件夹中,并将裁剪后的图像保存到“analysis/crops”文件夹中
(ii)训练和应用Ilastik像素分类器。可以在https://zenodo.org/record/7997296上访问在提供的示例数据上预训练的Ilastik像素分类器。
打开Ilastik并按照以下步骤操作:
• 创建一个新的“Pixel Classification”(像素分类)项目
• 在“Input Data”(输入数据)下,点击“Add New…”(添加新…)>“Add separate Image(s)”(添加单独的图像),并选择“analysis/crops”文件夹中的所有.h5文件
• 特征选择:我们一般建议选择与σ ≥ 1的所有特征
• 通过基于三类(标签1为“细胞核”,标签2为“细胞质”,标签3为“背景”)标记像素来训练Ilastik分类器。使用“Live Update”(实时更新)视图观察三类的预测结果。通过点击“Uncertainty”(不确定性)旁边的眼睛符号来观察不确定性,这将显示分类不确定性最高的区域。这些区域需要重新标记
▲ 关键提示:为了在分割过程中分离单个细胞,建议在两个靠得很近的细胞核之间绘制一条“细胞质线”,并用“细胞质”圈住细胞核。不应标记图像的大片区域,而应跨不同细胞和组织区域相对稀疏地放置像素标签。当在细胞核/细胞质和细胞质/背景边界处不确定性最高,而在其他位置不确定性较低时,分类器训练良好。
• 分类器训练良好后,将预测结果导出为无符号的16位TIFF图像
• 通过选择“analysis/ilastik”文件夹中的所有_s2.h5文件来批量处理所有文件。此步骤将生成包含图像分割像素概率的三通道(细胞核、细胞质、背景)图像
(iii)分割单个细胞。
将‘resources/pipelines/2_segment_ilastik.cppipe’流程导入CellProfiler,并执行以下步骤:
• 将‘analysis/ilastik’文件夹拖放到“Images”(图像)窗口中
• 在“Output Settings”(输出设置)中,将“Default Output Folder”(默认输出文件夹)调整为‘analysis/cpout’
以下步骤将作为流程的一部分自动执行:
• 读取像素概率,将其缩小以匹配初始图像的尺寸,并拆分为三个通道
• 将核通道和细胞质通道相加,形成一个单一通道
• 根据指定的面积阈值对细胞核进行分割和过滤
• 通过扩展核分割掩码到全细胞概率的边缘来分割整个细胞
• 将分割掩码作为16位单通道TIFF图像写入到‘analysis/cpout/masks’文件夹中
• 将缩小后的像素概率图像作为16位三通道TIFF图像写入到‘analysis/cpout/probabilities’文件夹中
◆ 故障排除
单细胞数据提取
● 选项A耗时5分钟,选项B耗时10分钟
- 从分割后的图像(即多通道图像和相应细胞掩码的配对)中提取空间分辨的单细胞数据。
(A) 基于深度学习的工作流程
(i) 对于每张图像,提取每个细胞和标记物的平均像素强度。得到的细胞水平的强度值以单独的CSV文件存储(每张图像一个文件):
1
steinbock measure intensities
(ii) 对于每张图像,提取每个细胞的形态学特征(面积、质心、长轴/短轴长度、默认离心率;若要计算其他形态学特征,请参阅steinbock在线文档)。得到的细胞水平特征以单独的CSV文件存储(每张图像一个文件):
1
steinbock measure regionprops
(iii) 在每张图像中,检测空间上接近的细胞。得到的空间细胞图(框3)以单独的CSV格式有向边列表存储(每张图像一个文件):
1
steinbock measure neighbors --type expansion --dmax 4
框3
空间细胞图
空间细胞图(也称为“邻域图”)是有向或无向的图(网络),其中细胞对应于节点,空间上邻近的细胞通过边相连。空间细胞图可用于分析细胞表型的空间共现情况(步骤31–35)。存在多种类型的空间细胞图;此处展示的是通过像素扩展构建的无向空间细胞图,在这种图中,仅当细胞在各方向上最多扩展4个像素后“接触”时,才认为它们是相邻的。
(B)基于像素分类的工作流程
(i)测量细胞特征。
“resources/pipelines/3_measure_mask.cppipe”流程用于测量单细胞和单图像特征。将其导入CellProfiler并执行以下步骤:
• 将“analysis/cpout”文件夹拖放到“Images”(图像)窗口中
• 在“Output Settings”(输出设置)中,将“Default Output Folder”(默认输出文件夹)调整为“analysis/cpout”,并将“Default Input Folder”(默认输入文件夹)调整为“analysis/cpinp”
以下步骤是该流程的一部分:
• 以16位灰度图像的形式读取分割掩码,并在保留其原始标签的同时将其转换为对象
• 通过定义的距离扩展掩码来检测每个细胞的邻居
• 提取每个细胞的完整堆栈通道强度和形状特征
• 提取每个细胞的概率堆栈通道强度
• 提取每幅图像的完整堆栈和概率堆栈强度
• 将细胞、图像和实验数据保存为CSV文件。单细胞强度值通过对应于位深度的缩放因子进行缩放。对于本方案中使用的16位无符号整数图像(uint16),这些值除以2^16 - 1 = 65,535
• 将空间细胞图导出为包含边列表的CSV文件
◆ 故障排除
单细胞和空间数据分析
本节中的命令必须在R中执行,RStudio软件可以简化执行过程。
数据读取
● 耗时5分钟
- 使用imcRtools包将空间分辨的单细胞数据读入R。在本方案的其余部分,我们将继续使用下面(A)中读入的steinbock生成的数据。
(A)读取使用steinbock工具包生成的数据
1 | library(imcRtools) |
(B)读取CellProfiler生成的数据
1 | spe2 <- read_cpout("path/to/analysis/cpout/") |
▲ 关键操作:调整路径以指向生成的空间分辨单细胞数据所在的位置。
- 在读取单细胞数据后,需要对SpatialExperiment对象(框4)进行进一步处理。首先,根据图像名称和细胞标识符设置列名。患者标识符和ROI标识符以及癌症类型也保存在对象中,这些信息可以从提供的“data/sample_metadata.csv”文件中读取。为了后续方便访问,选择了包含生物变异的通道。最后,使用1作为共因子,对每个通道和细胞的平均像素强度进行arsinh变换。
1 | library(tidyverse) |
▲ 关键提示 与通常使用5作为共因子的arsinh变换的CyTOF数据集相比,IMC数据集的强度通常要低得多。在这里,我们使用1作为共因子,这是对信号与噪声之间阈值的粗略估计。之前已有类似共因子值用于IMC数据9,11和MIBI数据64,65的分析。然而,对于不同类型的数据或信号强度更高/更低的IMC数据集,需要调整这个共因子。虽然这通常是根据定性标准手动完成的,但最近已有基于细胞类型特异性标志物共表达来启发式优化共因子值的系统方法被提出66。另外,也可以在此阶段对数据应用其他数据处理方法,包括Jackson等人8所使用的对数/平方根变换和数据截断。
- 使用cytomapper包25将多通道图像作为CytoImageList容器读入。
1 | library(cytomapper) |
▲ 关键提示 在使用steinbock工具包时,面板文件、图像和单细胞数据中的通道顺序是相同的。但是,当使用其他框架(如基于像素分类的分割方法)时,在设置通道名称时需要格外小心。
- 将分割掩码作为CytoImageList容器读入。
1 | masks <- loadImages("data/steinbock/masks/", as.is = TRUE) |
▲ 关键提示 除非另有配置(见“故障排除”),否则steinbock工具包会将分割掩码导出为16位图像。为了正确读取16位图像,需要设置‘as.is = TRUE’参数。在读取32位图像时,应使用默认参数设置(‘as.is = FALSE’)。
◆ 故障排除
框4
R中的单细胞数据容器
单细胞数据可以以SpatialExperiment(默认)或SingleCellExperiment对象17,28的形式读入。每个通道和细胞的平均像素强度存储在counts槽中。列代表细胞,行代表通道。与单个细胞相关的元数据存储在colData槽中。在初始图像处理后,这些元数据包括标签(ObjectNumber)、面积和每个细胞的形态学特征。此外,sample_id条目存储了从中提取每个细胞的图像名称。该工作流程支持SpatialExperiment和SingleCellExperiment对象。这些数据对象之间的主要区别在于存储所有细胞空间位置的方式。对于SingleCellExperiment容器,位置存储在colData槽中,而SpatialExperiment容器则将它们存储在spatialCoords槽中。steinbock等工具生成的空间细胞图被读入SpatialExperiment(或SingleCellExperiment)对象的colPair槽中。最后,与通道相关的元数据存储在rowData槽中。
- 对于下游的可视化和分析任务,需要在存储多通道图像和分割掩码的CytoImageList对象中添加额外的元数据。在这里,通过sample_id条目将单个图像、分割掩码和空间实验(SpatialExperiment)对象中的条目进行匹配。
1 | # 从图像名称中提取患者ID |
溢出校正(IMC特有)
● 耗时:20分钟
在IMC^(24)、MIBI^(67)和CyCIF^(68)等多重免疫荧光成像中,观察到两个通道之间存在低信号溢出(也称为串扰)。溢出被定义为在主要通道中可以检测到相邻通道信号的一小部分。由于溢出与相邻通道的信号成线性关系,因此可以通过之前描述的补偿方法^(24)进行校正。此处介绍的溢出校正是IMC特有的,如果假设已采用其他溢出最小化/校正方法,则在使用其他多重成像技术生成的数据时,可以跳过以下部分的步骤。
▲ 重要提示:如果核和细胞质通道相邻,也可以在图像分割之前进行溢出校正,以避免偏差。在这种情况下,需要将溢出校正后的图像写入“img”文件夹以供steinbock重新处理。然而,基于深度学习的分割方法对相邻通道之间的微量溢出不太敏感,而基于像素分类的分割方法则学会区分真实的核/细胞质信号和溢出信号。尽管如此,还是建议通过视觉检查确保用于分割的通道中的溢出量最小。
- 根据Chevrier等人24的研究,从溢出玻片中读取数据进行通道间溢出校正。创建和获取溢出玻片的实验过程可见补充说明2。根据CATALYST R/Bioconductor包69的建议,使用5作为辅因子对像素强度进行arsinh变换。
1 | sce <- readSCEfromTXT("data/compensation/") |
▲ 重要提示:提供的溢出玻片数据是专门为此数据集获取的,不能应用于其他数据集。因此,必须为每组使用的抗体-金属螯合物以及理想情况下为每个实验获取一个溢出玻片。
- 通过可视化每个通道和点样金属的中位像素强度来评估溢出数据的质量(图6a)。
1 | plotSpotHeatmap(sce) |
▲ 重要提示:评估每个通道和点样金属的中位像素强度有两个目的:首先,较小的中位像素强度(~200个计数)可能会妨碍对通道溢出的稳健估计。其次,每个点样金属(行)应在其对应的通道(列)中显示出最高的中位像素强度。
◆ 故障排除
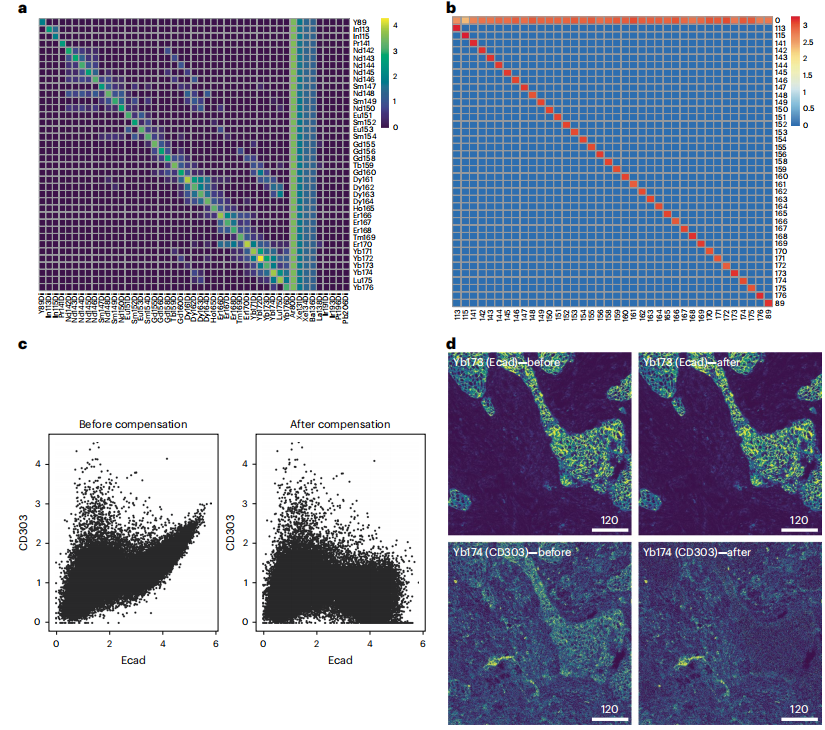
a,热图可视化每个通道(列)和点样金属(行)的log10转换后的中位像素强度。
b,热图可视化正确和错误分配给每个点样金属的像素的log10转换后的计数。x轴表示点样金属,y轴表示每个像素被分配到的金属。
c,通过散点图中可视化相邻通道(CD303:Yb174;Ecad:Yb173)的共检测标记强度,评估单细胞溢出校正的有效性。
d,图像显示了由于相邻通道溢出而产生的伪影信号。溢出校正消除了这些伪影。
- (可选)执行像素分箱以增加中位像素强度。这仅在像素强度过低(中位值低于约200计数)时才需要。
1 | sce2 <- binAcrossPixels(sce, bin_size = 10) |
- 过滤错误分配的像素。以下步骤使用CATALYST包提供的函数对像素进行“去条形码化”处理。基于所有通道的强度分布,将像素分配给其对应的条形码,这里即为已知的金属点。此过程会识别出无法稳健分配给点样金属的像素。此类像素可视为“噪声”、“背景”或“伪影”,并应在溢出估计之前去除。需要指定点样通道(bc_key)。
像素去条形码化的一般工作流程如下:
• 为每个像素分配一个初步的金属质量
• 对于每个像素,估计正像素集和负像素集之间距离的截断参数
• 应用估计的截断值来识别真正的正像素
1 | library(CATALYST) |
- 观察每个点正确和错误分配的像素数量。图6b中的热图描绘了对角线上分配给每个点的像素数量、非对角线上错误分配的像素以及第一行中无法分配给特定点的像素。对于当前数据集,所有像素要么被分配给相应的点,要么被标记为未分配。在以下代码块中,我们观察到有10%到55%的像素无法分配给其对应的点,这是可以预料的,因为点样抗体在溢出玻片上的空间分布可能是异质性的。在filterPixels函数中,minevents参数指定了正确分配的像素集被排除在溢出估计之外的阈值。correct_pixels参数指示是否将分配给除点样质量以外的质量的像素排除在溢出估计之外。默认值通常足以进行足够的像素过滤;但是,如果每个点测量的像素非常少(~100),则需要降低minevents参数的值。
1 | library(pheatmap) |
◆ 故障排除
- 使用CATALYST包计算和存储溢出矩阵。
1 | sce <- computeSpillmat(sce) |
- 使用CATALYST包执行单细胞数据补偿。compCytof函数使用先前估计的溢出矩阵直接在单细胞强度上校正通道间的溢出。CATALYST包提供的isotope_list变量需要扩展为包含不在此列表中的同位素。通过比较校正前后相邻通道(例如Yb173和Yb174)的标记强度可视化,可以评估溢出校正的有效性(图6c)。
1 | library(dittoSeq) |
- 对多通道图像进行通道间溢出校正。为此,需要对先前计算得到的溢出矩阵进行调整,仅保留存储在多通道图像中的通道。通过可视化相邻通道,可以评估溢出校正的效果(图6d)。
1 | # Use mass tags as channel names |
质控
● 时间:10分钟
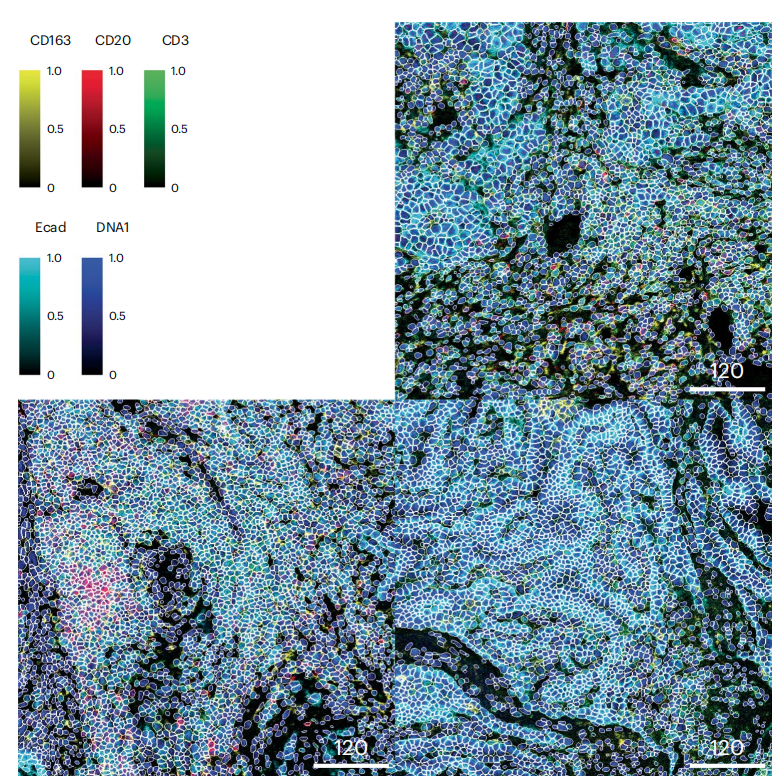
- 在组合图像上勾勒细胞轮廓,以便对分割质量进行视觉评估(图7)。为了可视化,我们从三组图像中选取子集,并在通道归一化后,在组合图像上勾勒出所有细胞的轮廓。
1 | set.seed(20220118) |
▲ 关键信息:这种可视化方法有助于识别图像中分割错误的细胞。建议可视化多张图像并放大以评估分割质量。如果单个细胞核由其各自的细胞掩膜勾勒出来,而没有明显的细胞聚集或分裂现象,则图像分割足够准确。如果测量的标志物定位于细胞核内或在细胞内相对均匀分布,则仅对细胞核进行分割,以减少来自相邻细胞的侧向溢出。这可以选择性地与steinbock utils的expand命令(参见steinbock在线文档)结合使用,以略微扩大细胞掩膜捕获的细胞区域11,70。
◆ 故障排除
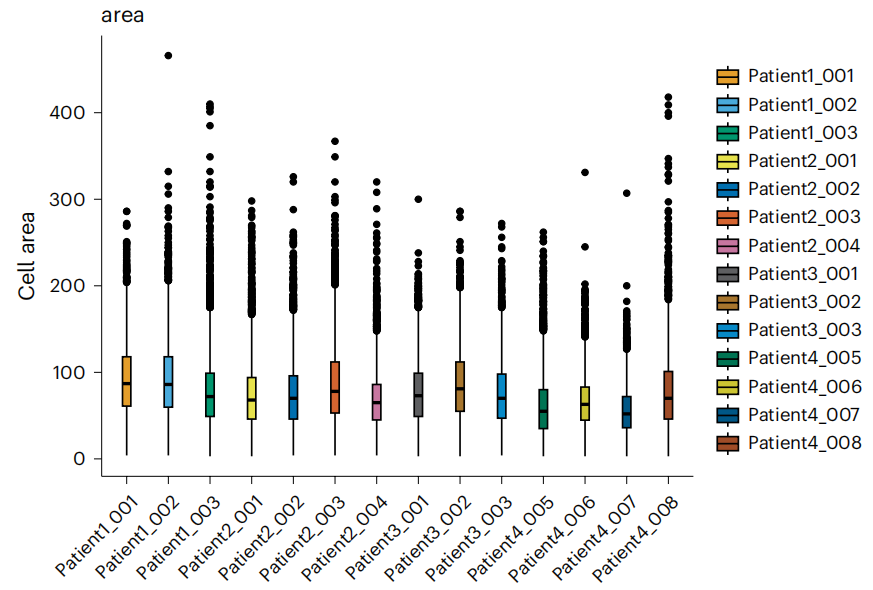
- 可视化细胞面积的分布(图8)并过滤掉小细胞。
1 | dittoPlot(spe, var = "area", group.by = "sample_id", plots = "boxplot") + ylab("细胞面积") + xlab("") |
▲ 关键信息:组织切片的多重成像通常不会捕获完整的细胞,而是测量细胞的切片。因此,可能无法准确代表相应细胞的小细胞切片应从分析中排除。所选阈值特定于数据集,应通过可视化细胞面积的分布进行微调。
◆ 故障排除
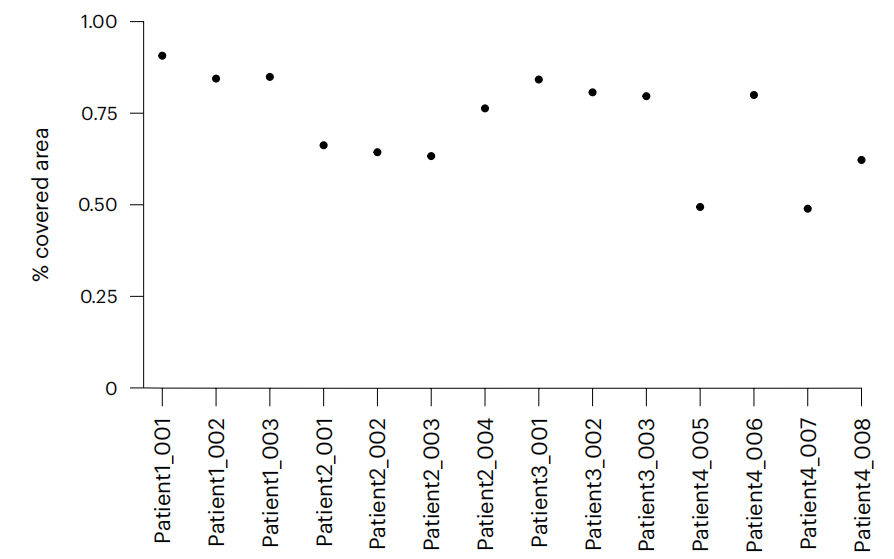
- 可视化每幅图像中细胞覆盖的图像区域(图9)。
1 | # 计算细胞覆盖的图像面积的比例 |
▲ 关键信息:在选择致密组织区域进行采集时,我们预期图像像素的大部分将被细胞覆盖。然而,由于稀疏组织结构(如血管)的存在或组织丢失,覆盖比例可能会降低。
◆ 故障排除
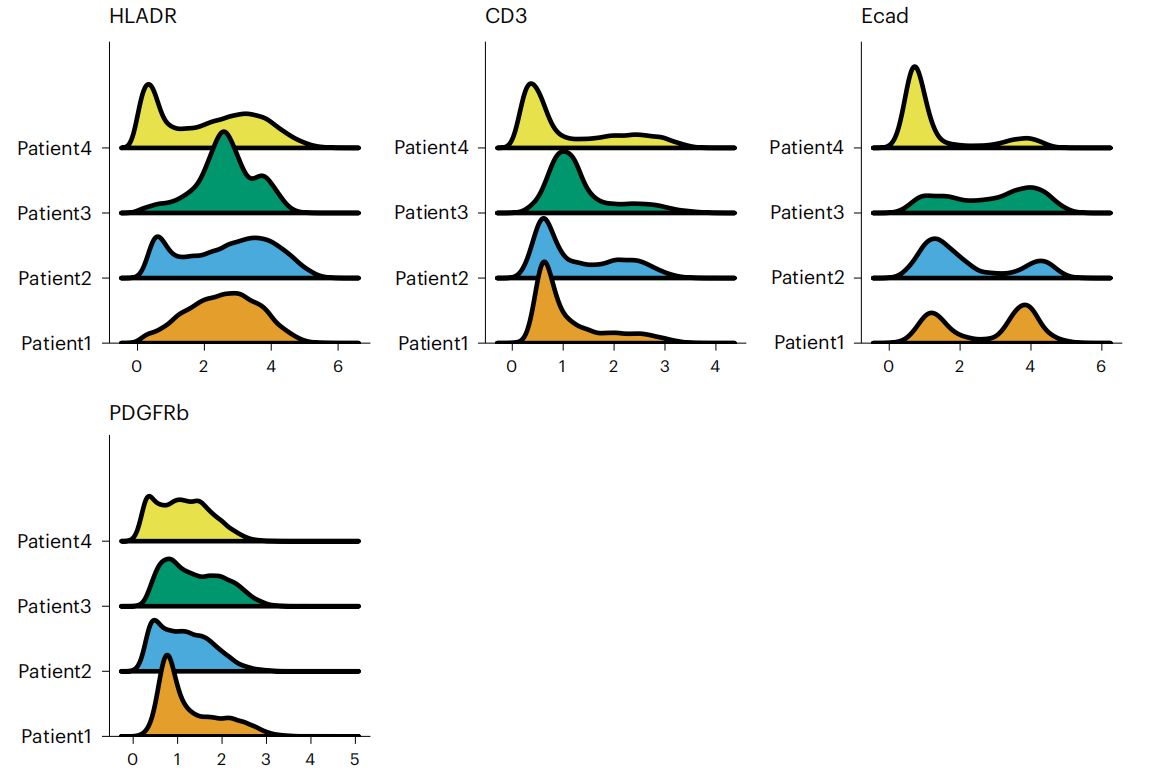
- 可视化选定标志物在样本间的染色差异。结合低维细胞可视化(步骤26),图10显示了样本间标志物表达的差异。
1 | multi_dittoPlot(spe, vars = c("HLADR", "CD3", "Ecad", "PDGFRb"), group.by = "patient_id", plots = c("ridgeplot"), assay = "exprs") |
- 可视化单细胞的低维嵌入。这里,我们使用scater包58来计算均匀流形近似和投影(UMAP)嵌入,并在低维空间中可视化细胞(图11a)。
1 | library(scater) |
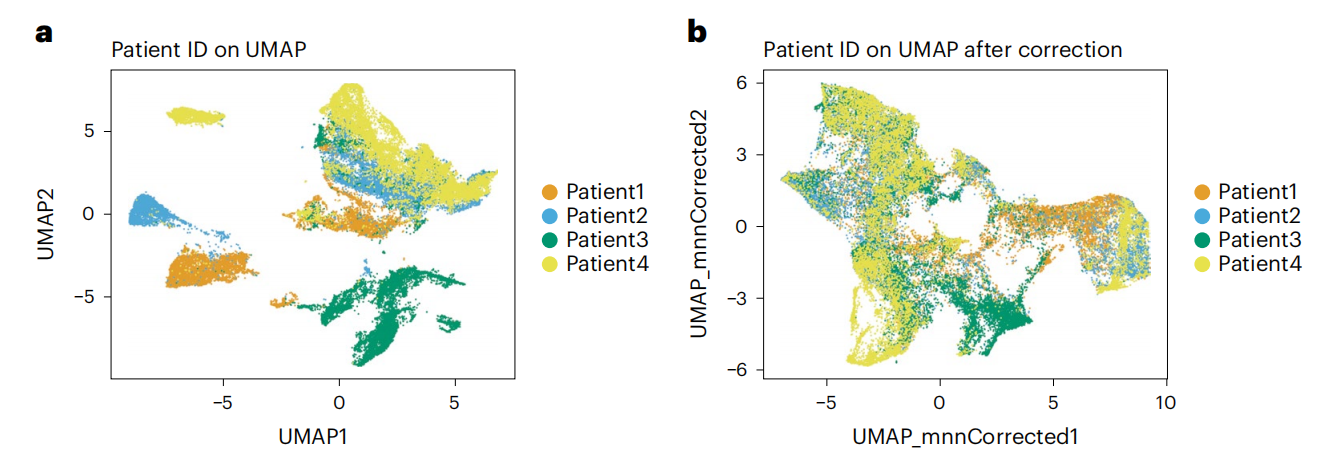
▲ 关键信息:在UMAP可视化(步骤25-26,图10和11a)中,标志物分布的差异或非重叠样本可能表明样本间的染色差异或生物学差异。当样本处理方式不同或使用不同批次的抗体混合物进行染色时,可能会出现染色差异。
◆ 故障排除
- 执行批次校正以消除样本间的差异(图11b)。这里,我们使用batchelor包的fastMNN方法30。在以下代码块中,为了可重复性,在设置种子后依次执行非确定性的fastMNN和runUMAP函数。
1 | library(batchelor) |
▲ 关键信息:需要视觉评估批次校正的准确性,以估计是否除了技术变异外还消除了生物变异。这可以通过根据已知在某些细胞表型中表达的标志物对UMAP嵌入进行着色来实现。这些标志物应在UMAP嵌入中聚类的细胞中表达。可以使用其他批次效应校正方法来改进数据整合71,72。
◆ 故障排除
细胞表型分析
● 时间:选项A需30分钟,选项B需1-2小时(更大数据集需要更多时间)
- 定义细胞表型。为此,可以对单细胞进行聚类(A)或通过分类进行表型分析(B)。
(A)通过聚类进行细胞表型分析
使用bluster和scran R/Bioconductor包59中的函数进行基于图的聚类。另外,也可以使用其他方法,如phenograph53或FlowSOM73对单细胞进行聚类。
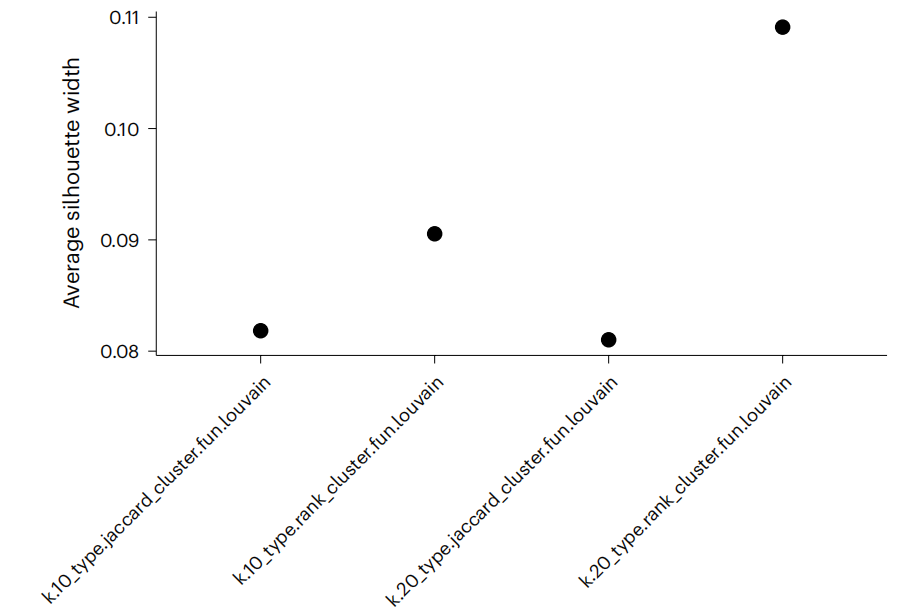
(i)在批次校正后的细胞嵌入上估计基于图的聚类的最佳聚类参数。我们遍历聚类参数的所有可能组合(图12),包括要考虑的最近邻数(k)和边权重方法(类型)。我们保持社区检测算法(cluster.fun)固定,因为Louvain方法74是基于图的聚类中最常用的算法之一。为了评估聚类的稳定性,我们计算所有细胞的平均轮廓宽度,并选择平均轮廓宽度最高的聚类参数组合。
1 | library(bluster) |
▲ 关键信息:对于每个数据集,应独立进行参数估计。对于大型数据集,该函数运行时间较长,为了近似处理,可以对数据集进行子采样以加快聚类遍历的速度。
(ii)基于所选参数,使用基于图的算法对细胞进行聚类。如前所述,将参数设置为k=20和类型“rank”应该能得到分离度较好的聚类。然后,将聚类标识符保存在SpatialExperiment对象中。
1 | library(scran) |
◆ 故障排除
(iii)为了根据各聚类中包含的细胞表型对其进行注释,可以以热图的形式可视化每个聚类的标志物表达(图13)。为了可视化目的,从数据集中随机选择了2000个细胞。
1 | library(viridis) |
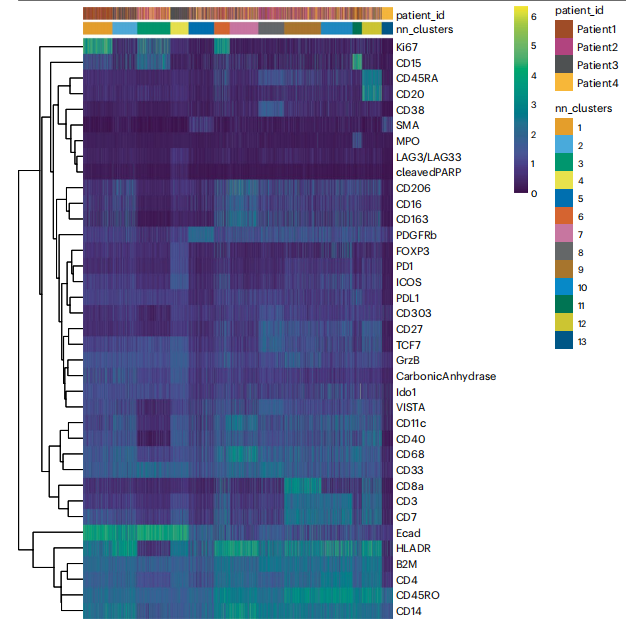
(iv)在观察到聚类特异性表达模式后,可以对各个聚类进行注释。例如,聚类12显示出B细胞标志物CD20的高表达,因此可以标记为“Bcell”。
1 | cluster_celltype <- recode(spe$nn_clusters, |
(B)基于分类的细胞表型分析
(i)使用cytomapper包中包含的cytomapperShiny函数,根据标志物的表达来界定单个细胞的表型。对于每张图像,细胞根据其标志物表达的层次结构进行界定,以定义预期的细胞表型。然后,将界定的细胞以轮廓的形式显示在伪彩色合成图像上。一旦正确标记了细胞,就可以将它们作为仅包含选定细胞的SpatialExperiment对象下载。在下载时,可以指定细胞标签,该标签存储在colData插槽的cytomapper_CellLabel条目中,以便稍后用于训练分类器(步骤28B(iv))。
1 | cytomapperShiny(object = spe, mask = masks, image = images_comp, |
▲ 关键信息:由于分割不完全,侧向溢出会导致某些标志物的表达水平不正确。因此,我们建议仅对表达其特定细胞表型标志物的细胞进行界定。
(ii)读取包含界定细胞的文件,并将它们合并成一个SpatialExperiment对象。
1 | library(SingleCellExperiment) |
(iii)移除被多次标记的细胞,并重新分配肿瘤细胞。由于采用多步骤标记方法,一些细胞可能被标记了多次。在细胞同时被标记为肿瘤细胞和免疫细胞的情况下,我们保留免疫细胞标签,因为这些细胞很可能是驻留在肿瘤内的免疫细胞。所有其他被多次标记的细胞都被移除。最后,将标签存储在主要的SpatialExperiment对象中。我们首先定义一个辅助函数,该函数返回仅被标记一次的细胞的标签。
1 | filter_labels <- function(object, |
接下来,应用此函数来检索所有仅被标记一次的细胞。
1 | labels <- filter_labels(concat_spe) |
再次对所有细胞应用该函数,同时排除“Tumor”标签。这种方法可以识别那些既被标记为肿瘤细胞又被标记为免疫细胞的细胞。
1 | cur_spe <- concat_spe[,concat_spe$cytomapper_CellLabel != "Tumor"] |
最后,将两组细胞标签合并并存储在SpatialExperiment对象中。
1 | final_labels <- c(labels, non_tumor_labels[additional_cells]) |
(iv)为未标记细胞的表型分类训练一个随机森林分类器。首先,将细胞分为标记细胞和未标记细胞。然后,我们对标记细胞进行75/25的划分,以分别选择训练集和测试集。基于训练集,我们进行五折交叉验证来调整随机森林模型的参数。在以下代码块中,为了可重复性,在设置随机种子后,依次执行了非确定性的createDataPartition函数和train函数。
1 | library(caret) |
◆ 故障排除
(v)通过计算测试集的混淆矩阵来评估分类器的性能。confusionMatrix函数将预测的细胞标签与真实的细胞标签进行比较,并计算多个性能指标。期望每个细胞表型标签都具有高灵敏度和高特异性。
1 | # Select the arsinh-transformed counts of the test data |
(vi)预测未标记细胞的标签。对于最高类别概率低于40%的细胞,将其标记为“未知”。
1 | # Select the arsinh-transformed counts of |
▲ 关键信息:将细胞标记为未定义的阈值需要根据每个数据集进行调整。当预测更多的细胞类别时,阈值可能会降低;而当预测较少的类别时,阈值可以相应提高。
- 在UMAP嵌入上可视化细胞表型和注释后的聚类标签,以定性评估细胞表型(图14)。
1 | p1 <- dittoDimPlot(spe, var = "celltype", |
- 以热图的形式可视化每个细胞表型和每个注释后的聚类的平均标志物表达(图15)。
1 | library(scuttle) |
▲ 关键信息:我们期望单个细胞表型能够显示出其特异性标志物的最高平均表达。此外,在如UMAP这样的低维嵌入中,细胞表型应能在视觉上被区分开来。
◆ 故障排除
空间分析
● 预计时间:20分钟
本方案介绍了一系列空间分析方法,旨在研究细胞在组织环境中的分布情况。如需概览,请见图16。
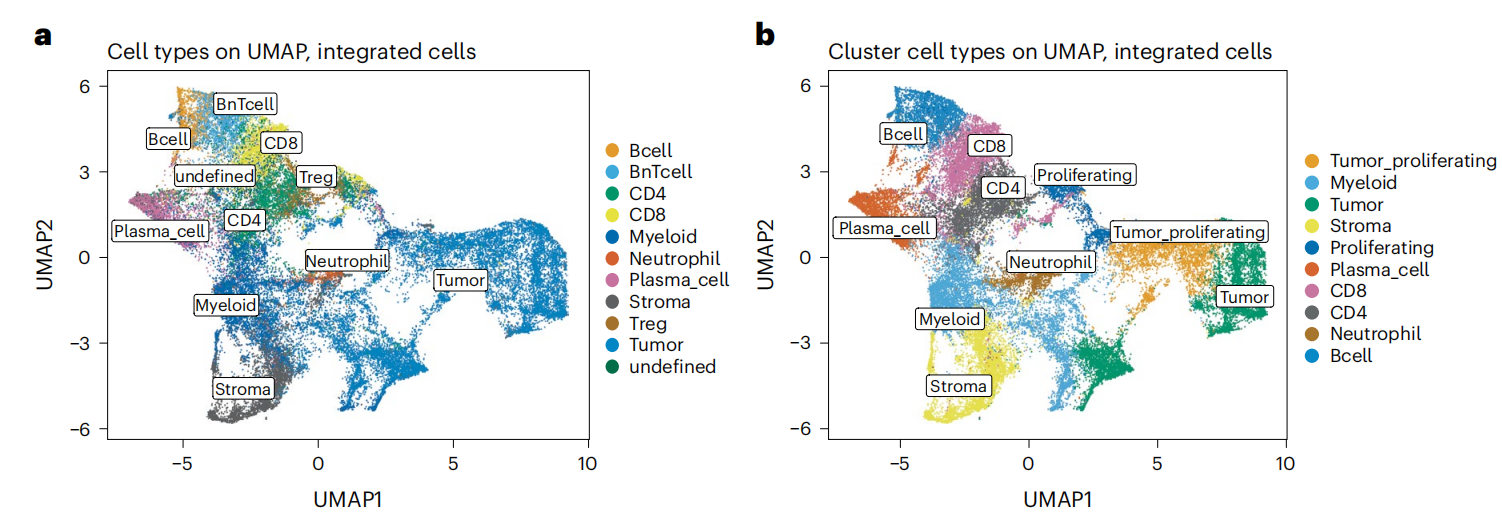
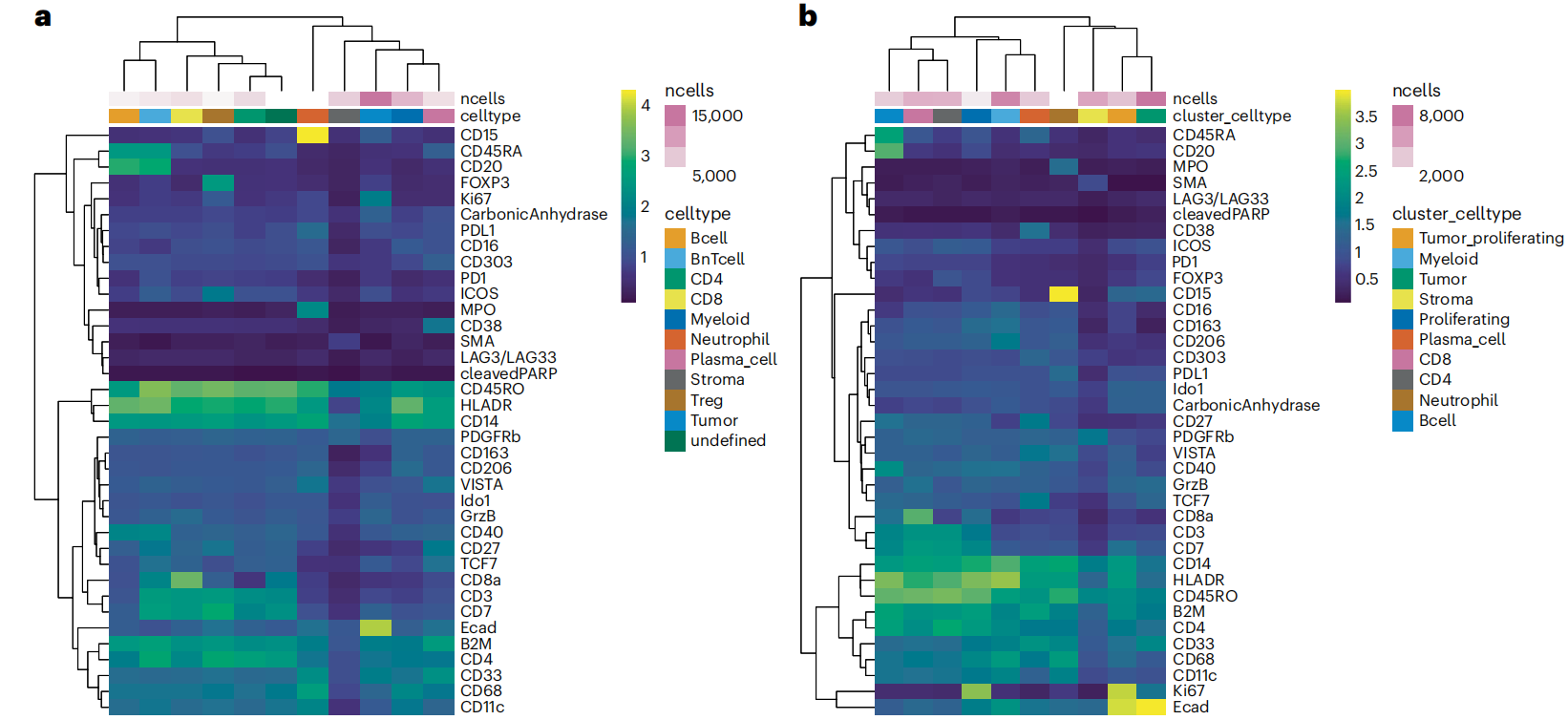
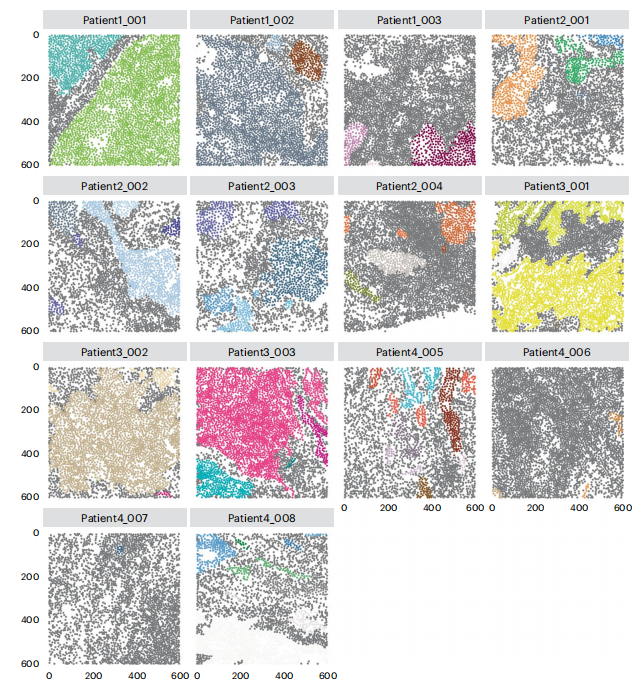
- 使用Jackson等人提出的空间群落分析方法8(图17),并借助imcRtools软件包提供的detectCommunity函数。该方法通过先前构建的空间细胞图,仅根据细胞在组织中的位置对细胞进行分组。我们分别对肿瘤细胞和非肿瘤细胞进行了群落检测。
1 | # Define if cells are part of the tumor or stroma |
在检测到空间群落之后,可以进行一系列下游分析。这些分析包括(i)计算用于元聚类的每个群落的细胞表型比例,(ii)识别样本间的共享群落以进行比较分析,以及(iii)计算单个群落的大小以估计组织分隔。
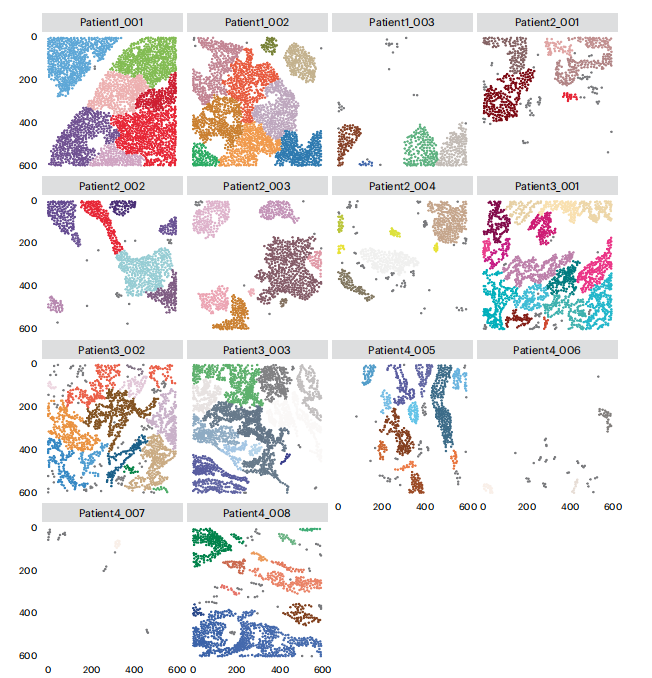
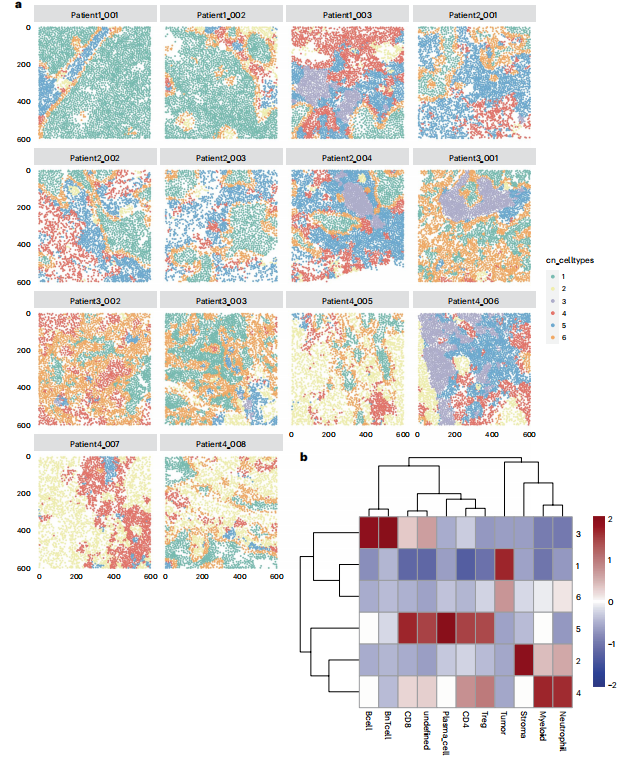
- 按照Schürch等人10和Goltsev等人5的提议执行拷贝数(CN)分析(图18)。拷贝数是具有特征细胞表型组成的组织区域,它们代表了独特的局部生物过程和相互作用的位点。我们首先基于细胞的质心构建一个空间20近邻图,与先前使用steinbock构建的空间细胞图相比,该图包含了更多的邻居。然后,使用imcRtools包的aggregateNeighbors函数计算每个细胞在其20近邻中的细胞表型比例。接着,使用k均值聚类对细胞进行聚类以检测拷贝数。最后,可以根据细胞的拷贝数对其进行空间可视化和着色,并计算每个拷贝数的细胞表型比例。在图18中,拷贝数3代表B细胞的空间聚集,而拷贝数1主要包含肿瘤组织。
1 | # Construct a 20-nearest neighbor graph |
▲ 关键步骤:与第28步类似,应通过参数扫描来估计k的最优值。但是,如果事先了解要检测的组织结构,则可以相应地设置k值。或者,也可以根据细胞邻域内细胞的平均标志物表达来对细胞进行聚类。此外,lisaClust R/Bioconductor包还提供了检测拷贝数(CNs)的其他策略75。
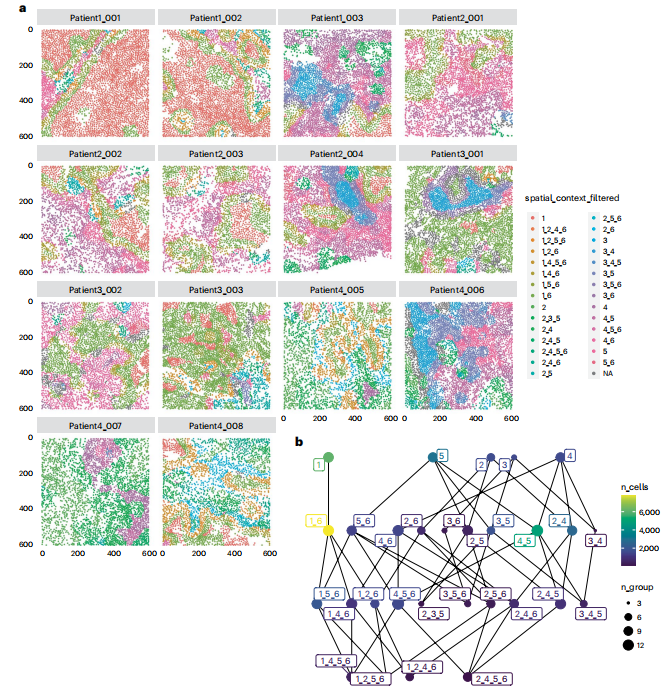
- 按照Bhate等人19的提议执行空间背景(SC)分析(图19)。空间背景建立在拷贝数(CNs)的概念之上,是拷贝数的局部生物过程相互作用并发生特殊生物事件的区域。我们构建了第二个k近邻图,其中k值较大(k=40),以包含可在生物信号交换的长度尺度上的细胞。对于每个细胞,aggregateNeighbors函数计算其40个最近邻中拷贝数的比例。detectSpatialContext函数将拷贝数比例从高到低排序,并将每个细胞的空间背景分配为累积超过用户定义阈值(此处为0.9)的最小拷贝数组合。在过滤检测到的空间背景后,我们可以对其进行空间可视化,并将空间背景相互作用表示为分层图。我们观察到,SC 1(以肿瘤为主)、SC 1_6(肿瘤与肿瘤-间质界面)和SC 4_5(浆细胞/T细胞和髓系细胞/中性粒细胞界面)是该数据集中最常见的空间背景。
1 | # Construct a 40-nearest neighbor graph |
- 按照Hoch等人11的提议执行斑块检测分析(图20)。imcRtools包的patchDetection函数检测感兴趣细胞的完全连接成分,在每个成分周围构造凹包,并将此凹包扩展以包含相邻细胞。在下文中,我们检测包含至少十个细胞的连接肿瘤成分,并稍微扩展凹包以包含斑块内的细胞。
1 | spe <- patchDetection(spe, |
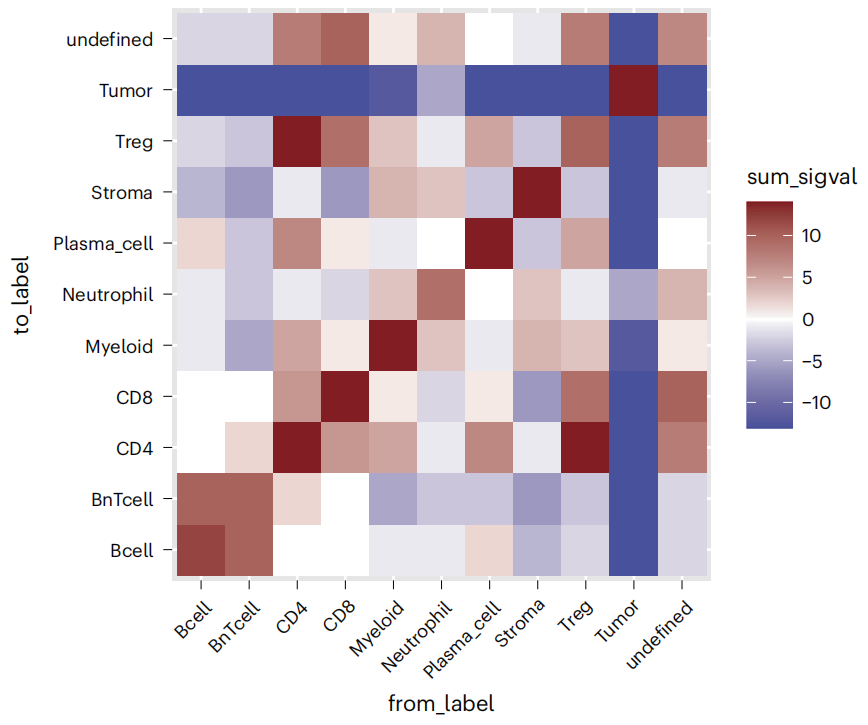
- 按照Schapiro等人18的提议执行相互作用分析(图21)。该方法检测与细胞表型的随机分布相比表现出更强(“相互作用”)或更弱(“避免”)共定位的细胞表型对。使用先前构建的空间细胞图(此处为使用steinbock创建的图),imcRtools包的testInteraction函数计算每幅图像中每对细胞表型的平均相互作用次数,并将其与通过随机置换所有细胞标签获得的经验空分布进行比较。返回的数据框包含每幅图像中每对细胞表型的一个条目,指示经验P值和统计显著性(相互作用:1;无显著性:0;避免:-1)。这些显著性值可以跨所有图像求和,并以热图的形式可视化。我们观察到,肿瘤细胞大多被分隔开,并与其他细胞类型相互避免,而调节性T细胞则位于CD4+ T细胞和CD8+ T细胞旁边。
1 | library(scales) |
◆ 故障排除
参考
- Windhager J , Zanotelli V R T , Schulz D ,et al.An end-to-end workflow for multiplexed image processing and analysis[J].Nature Protocols, 2023, 18(11).DOI:10.1038/s41596-023-00881-0.
加关注
 |
 |





